全基因组与宏基因组建库技术
引言
基因组学技术的快速发展为生命科学领域带来了革命性突破。全基因组测序(Whole Genome Sequencing, WGS)与宏基因组学(Metagenomics)作为两大核心分支,分别在单一生物体基因组解析和复杂微生物群落研究领域发挥着关键作用。本文将从技术原理、建库流程、应用场景及未来挑战等方面系统阐述这两项技术的科学价值与实践意义。
一、全基因组建库技术
1. 基本概念
全基因组测序是通过高通量测序技术对生物体的全部遗传信息进行解析,覆盖编码区(外显子)与非编码区(内含子),能够检测单核苷酸变异(SNP)、插入缺失(Indel)、拷贝数变异(CNV)等各类遗传变异 。与全外显子组测序(WES)相比,WGS无需靶向捕获,可提供更全面的基因组信息,尤其适用于新突变位点的发现 。

DNA双螺旋结构示意图
2. 技术流程
全基因组建库的核心步骤包括:
- 样本制备:从血液、组织等样本中提取高质量DNA,需避免降解 。
- DNA片段化:通过超声破碎或酶切法将DNA打断为200-500 bp的片段 。
- 末端修复与接头连接:修复DNA片段末端并添加测序接头,便于后续扩增与测序。
- 文库构建:通过PCR扩增富集目标片段,并进行质量评估(如片段长度分布检测) 。
- 测序与数据分析:基于新一代测序平台(NGS)生成原始数据,经比对、拼接和变异识别后获得基因组图谱 。
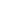
高通量测序仪工作流程示意图
3. 应用领域
- 医学诊断:WGS可识别罕见遗传病的致病突变,例如囊性纤维化与亨廷顿舞蹈症的基因诊断 。
- 肿瘤研究:通过分析肿瘤组织与正常组织的基因组差异,揭示驱动突变并指导靶向治疗 。
- 农业育种:在家畜(如猪、牛)中筛选与生长速度、抗病性相关的遗传标记,加速分子育种进程 。
二、宏基因组建库技术
1. 基本概念
宏基因组学通过直接测序环境样本中的全部微生物DNA,无需培养即可解析群落结构与功能 。其研究对象涵盖土壤、水体、人体肠道等复杂环境,能够发现未培养微生物的新基因与代谢通路 。
2. 技术流程
宏基因组建库的关键步骤包括:
- 样本处理:根据来源(如粪便、水体)进行过滤、离心以富集微生物细胞 。
- DNA提取:使用CTAB法或试剂盒获取混合DNA,需避免宿主DNA污染(如人类肠道样本) 。
- 片段化与文库构建:将DNA随机打断并连接测序接头,构建适用于短读长或长读长平台的文库 。
- 功能注释:通过KEGG、COG等数据库对基因功能进行分类,揭示群落的代谢潜力 。
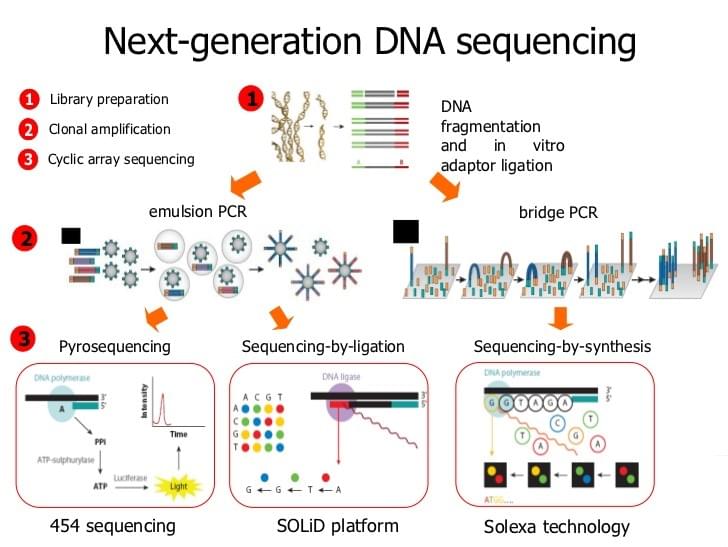
微生物DNA提取实验操作示意图
3. 应用领域
- 环境修复:分析污染土壤中的降解基因,指导生物修复策略 。
- 人类健康:解析肠道菌群与肥胖、糖尿病的关联,开发益生菌干预方案 。
- 农业创新:筛选促进植物生长的根际微生物,减少化肥使用 。
三、技术比较与协同应用
| 维度 | 全基因组测序 | 宏基因组学 |
|---|---|---|
| 研究对象 | 单一生物体基因组 | 微生物群落混合DNA |
| 技术难点 | 高覆盖度与长片段拼接 | 宿主污染控制与数据去卷积 |
| 数据分析 | 变异注释与致病性预测 | 物种分类与功能网络构建 |
| 应用场景 | 遗传病诊断、分子育种 | 环境监测、微生物组与疾病关联研究 |
二者的协同应用可推动多组学研究,例如结合宿主基因组与肠道菌群数据,解析基因-环境互作对疾病的影响 。
四、挑战与未来展望
1. 技术瓶颈
- 数据解读:WGS中非编码区变异的生物学意义尚不明确;宏基因组数据分析需解决物种注释错误率高的难题 。
- 成本与时效性:尽管测序成本下降,但大规模样本分析仍需要高性能计算资源支持。
2. 发展方向
- 长读长测序技术:提升复杂区域(如高度重复序列)的组装准确性 。
- 单细胞分辨率:在宏基因组学中实现菌株水平的分辨,揭示微环境异质性 。
- 人工智能整合:利用深度学习模型预测变异功能或微生物群落动态 。
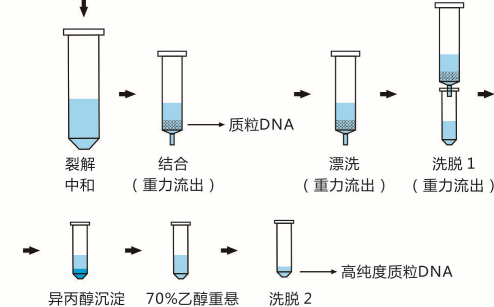
人工智能分析基因组数据示意图
结语
全基因组与宏基因组建库技术已成为生命科学研究的基石工具。随着测序技术的迭代与多学科交叉融合,这两项技术将在精准医学、生态保护、农业可持续发展等领域持续释放潜力,为人类探索生命奥秘提供更强大的技术支撑。
| 名称 | 货号 | 规格 |
| DNeasy PowerLyzer PowerSoil Kit (100) | 12855-100 | 100Test |
| DNeasy PowerSoil Pro Kit (50) | 47014 | 50Test |
| DNeasy PowerSoil Pro Kit (250) | 47016 | 250Test |
| DNeasy PowerSoil HTP 96 Kit (384) | 12955-4 | 384Test |