全转录组扩增(WTA)
一、单细胞测序的概念及特点
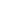
单细胞测序技术是指在单细胞分辨率层面,针对基因组、转录组等组学信息开展高通量测序分析的技术体系。尽管其单次实验样本量可扩展至数千乃至上万个细胞(具体规模依据样本类型与研究需求而定),但单细胞分析的核心价值并非单纯追求样本总量,而是通过解析单个细胞的遗传特征实现细胞异质性的精准解析。
为何需要单细胞分辨率的测序?
多细胞生物虽源于单个受精卵的增殖分化,但发育过程中细胞间会逐渐积累遗传与表观遗传差异,形成功能特化的细胞亚群。即便传统分类中同属某一细胞类型,其内部仍存在显著的分子异质性,这些差异正是决定细胞功能多样性的关键因素。
传统测序技术的局限性
传统测序技术聚焦于组织或细胞群的整体分析,所得数据为细胞群体的平均信号,无法捕捉细胞间的异质性特征。而单细胞测序通过解析每个细胞的基因表达谱,能够直接揭示细胞亚群的分子特征与发育轨迹,为理解细胞功能分化的分子机制提供了革命性工具。
二、单细胞检测策略
当前单细胞检测领域主要发展出两类技术路线:
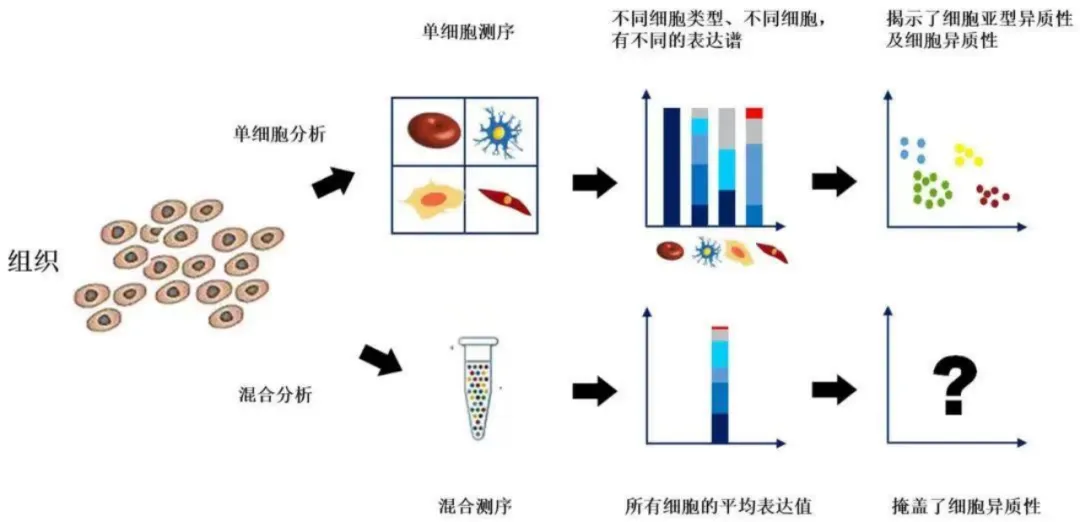
1. 基于物理分离的靶向检测策略
通过流式细胞术(适用于悬浮细胞样品)或激光显微切割(LCM)技术(针对组织切片样本)实现单细胞物理分离,继而构建测序文库并进行独立测序。
该方法存在固有局限性:检测通量低且成本随细胞数量呈线性增长,尤其面对临床样本中10⁹量级的细胞群体时,仅分析数十个细胞可能导致细胞异质性信息丢失和统计代表性不足,难以满足复杂组织的研究需求。
2. 基于分子标记的高通量检测策略
采用微流控技术将单细胞悬液与携带独特分子标签(barcode)的磁珠共同包裹在纳升级油滴中,通过逆转录反应使每个细胞的cDNA文库携带特异性barcode标记。最终通过混合建库和统一测序,借助生物信息学算法解码barcode实现单细胞数据区分。
该技术突破性地实现了单次实验完成数千细胞并行分析,显著提升了检测效率与经济性。其核心技术优势在于:
- 组合标记策略:通过10⁸量级的barcode组合确保细胞身份可追溯性
- 分子扩增偏倚控制:油滴微环境有效减少扩增竞争效应
- 数据解复用算法:开发专用计算框架实现高效信号解卷积
三、单细胞分离制备标准化流程(以肿瘤组织为例)
1. 单细胞悬液制备规范
(1)组织采集与活性维持
采用颈椎脱臼法快速处死实验动物,解剖过程需在低温环境(4℃)中操作,推荐使用预冷的RPMI-1640培养基(含10% FBS)进行组织冲洗,以最大限度保留细胞活力。
(2)组织解离优化策略
- 机械解离:根据下游分析需求(侧重细胞数量或质量)选择组织块体积,在冰浴条件下进行精细剪碎(建议终体积≤1 mm³)。
- 酶解消化:针对不同组织特性选择胶原酶/胰蛋白酶配比及消化时间(如肿瘤组织常用胶原酶IV 200 U/mL + DNA酶I 50 U/mL,37℃消化15-30 min),需通过台盼蓝染色实时监测细胞活性。
- 红细胞去除:对富血红素组织(如肝脾)采用NH₄Cl裂解液处理,需严格控温(37℃作用3 min后立即冰浴终止),重复裂解不超过2次以避免白细胞损伤。
2. 单细胞筛选技术比较
| 技术类型 | 作用原理 | 分辨率 | 通量 | 成本 | 适用场景 |
|---|---|---|---|---|---|
| 有限稀释法 | 梯度稀释至单细胞水平 | 低(群体水平) | 极低 | 低 | 教学演示,已逐渐被淘汰 |
| 显微微管操作 | 手动显微操控捕获 | 单细胞 | <10 cells/h | 高(人力成本) | 稀有微生物分离,临床样本不适用 |
| 激光捕获显微切割 | 激光脉冲切割靶细胞 | 单细胞 | 中等 | 中 | 临床病理样本,但易产生污染 |
| 微流控技术 | 流体动力学精准操控 | 单细胞 | 高(>1000/h) | 极高 | 高精度研究,受限于设备普及度 |
| 流式细胞分选 | 多参数荧光信号分选 | 单细胞 | 极高(>5000/h) | 中 | 主流方法,需平衡速度与细胞活性 |
技术细节补充:
- 流式细胞术优化:采用低流速(<5000 events/s)和分选模式(Purity模式)可显著降低细胞损伤,建议预实验确定目标细胞群FSC/SSC阈值。
- 微流控技术前沿:集成惯性聚焦或介电泳原理的新型芯片,已实现无标记单细胞分选,但需配套专用测序文库构建体系。
四、遗传物质提取与扩增技术
单细胞测序面临的核心挑战在于起始样本中遗传物质的极端稀缺性:典型哺乳动物细胞仅含约10 pg总RNA(其中mRNA占比仅0.1-0.5 pg),而深度测序要求纳克级DNA输入,这意味着需将原始核酸扩增10⁵-10⁶倍。目前,单细胞扩增技术主要分为全基因组扩增(WGA)和全转录组扩增(WTA)两大体系。
1. 全基因组扩增技术演进
-
简并寡核苷酸引物PCR(DOP-PCR)
通过随机引物介导的全基因组扩增,早期实现了单细胞DNA的指数扩增。但该方法存在显著缺陷:扩增覆盖度不均(基因组某些区域过度扩增,其他区域丢失),且易引入PCR偏差,已逐渐被淘汰。 -
多重置换扩增(MDA)
基于Φ29 DNA聚合酶的等温扩增技术,可在常温条件下完成扩增,避免高温导致的DNA损伤。该技术能保留长片段DNA(>10 kb),适用于单核苷酸多态性(SNP)检测和突变分析。但扩增均匀性不足,存在GC含量依赖的扩增偏好。 -
多次退火环状循环扩增(MALBAC)
第三代WGA技术,通过预扩增引物设计和线性扩增策略,显著提升基因组覆盖率和均匀性(>90%覆盖率,偏差系数<0.2)。特别适用于稀有样本(如循环肿瘤细胞)和高度异质样本(如发育早期胚胎细胞)的研究。
2. 全转录组扩增技术体系
单细胞转录组扩增需先提取mRNA并反转录为cDNA,再通过扩增获得检测信号。主流方法包括:
- PCR指数扩增:快速高效但易引入偏差
- 体外转录线性扩增:保真度高但步骤复杂
- Phi29聚合酶扩增:等温扩增且覆盖度优,但成本较高
技术选择原则:
- 研究基因结构变异→优先选择MALBAC
- 检测点突变或拷贝数变异→推荐MDA
- 转录组定量分析→需结合扩增偏差校正算法
| 名称 | 货号 | 规格 |
| Rhapsody反转录试剂盒 | 633773-1 | 1 Test |
| REPLI-g WTA Single Cell Kit (24) | 150063 | 24Test |
| REPLI-g Cell WGA & WTA Kit (12) | 150052 | 12Test |
| BD Rhapsody WTA Amplification Kit | 633801-BDRhapsody | 1Each |