单细胞RNA测序
前言
单细胞RNA测序(scRNA-seq)作为解析细胞异质性的革命性工具,已能实现单次实验对数千乃至上万个细胞的高通量分析,为复杂组织细胞图谱的绘制提供了技术支撑。然而,当前多种scRNA-seq技术平台在化学原理、扩增效率及数据产出特征上存在差异,其对于组织器官中细胞类型与功能状态的解析能力尚未形成系统性评估。近期《自然·生物技术》(Nature Biotechnology)刊发的跨国合作研究,通过13种主流scRNA-seq技术对同一组细胞样本进行平行分析,首次建立了该技术领域的系统性基准测试框架,为方法学优化与跨平台数据整合提供了关键依据。
实验设计
1. 样本设计原则
研究团队构建了多参数可控的细胞混合体系,以全面评估不同技术特性:
- 异质性梯度:包含高频主流细胞类型与稀有细胞亚群,模拟生物组织中细胞丰度分布
- 亚群分辨率:设计基因表达谱高度相似但存在细微差异的细胞亚群,检验技术对细微生物学差异的捕捉能力
- 谱系追踪标记:整合荧光蛋白标记的细胞系,建立细胞身份的金标准参照系
- 跨物种对照:引入人类、小鼠和犬类细胞,验证技术对不同物种的普适性
2. 实验流程及样本构成
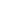
采用三物种五细胞系的混合体系(图1):
- 人类外周血单个核细胞(PBMC)(60%):代表高度异质性的原代细胞群体
- 小鼠结肠细胞(30%):体现组织特异性细胞组成
- HEK293T细胞系(6%,RFP标记):提供红色荧光蛋白标记的人类细胞对照
- NIH3T3细胞系(3%,GFP标记):绿色荧光蛋白标记的小鼠细胞系
- MDCK细胞系(1%,TurboFP650标记):远红外荧光蛋白标记的犬类细胞系
该混合样本按预设比例分配为13份,分别采用不同scRNA-seq技术方案进行处理。此设计通过控制变量法,在标准化实验条件下系统比较各技术平台的细胞检测灵敏度、基因表达定量准确性及细胞类型分辨率等核心性能指标。
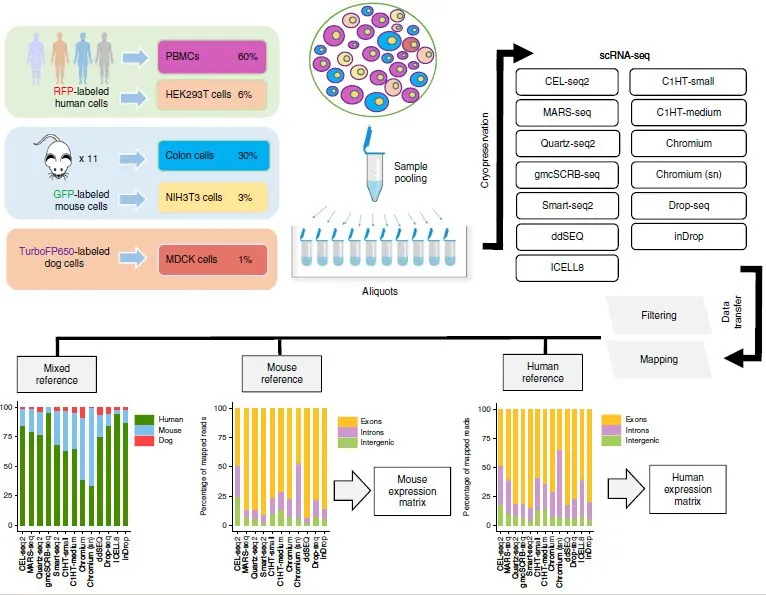 图1 实验流程
图1 实验流程
3. 扩增方法
本研究系统部署了13套实验方案,全面覆盖表1所列11类核心技术体系,构建起多元化的单细胞扩增方法矩阵
实验结果
1. 物种特异性检测效能与转录本特征
跨平台分析结果显示(图1),各技术方案对混合样本中人类细胞(63%-95%)和小鼠细胞(4%-34%)的捕获效率呈现显著差异:
- 人类细胞检测偏向性:10X Genomics的细胞核/细胞质分离方案显示人类细胞占比显著低于其他方法(P < 0.01),而gmcSCRB-seq与ICELL8系统则表现出更高的人类细胞捕获效率(P < 0.05)。
- 稀有细胞检测能力:犬源MDCK细胞(掺入量1%)的检出率在不同平台间波动显著(0%-9%),其中gmcSCRB-seq未检出该亚群,反映部分技术对低丰度细胞的检测灵敏度存在局限。
- 转录本完整性差异:由于细胞核内含有未剪接的pre-mRNA,10X细胞核测序方案捕获的内含子序列比例显著高于其他方法(中位数差异 > 3倍),表明不同技术体系在RNA分子保留特性上存在差异。
2. 基因检测通量与细胞类型特异性表达
对HEK293T细胞系、单核细胞及B细胞的定量分析(图2)揭示:
- 跨细胞系检测一致性:Quarts-seq2在所有细胞类型中均展现出最高的基因检出数(HEK293T细胞:~12,000 genes;单核细胞:~9,500 genes;B细胞:~8,200 genes),体现其卓越的转录组覆盖能力。
- 方法特异性表现:CEL-seq2在HEK293T细胞中表现优异(基因数 >11,000),但在单核细胞和B细胞中的检测效能仅次于Quarts-seq2而优于其他方法(基因数差异 > 1,000),表明不同技术对不同细胞类型的适配性存在差异。
- 灵敏度分层现象:Smart-seq2与Chromium系统在HEK293T细胞中显示中等检测能力(基因数 ~10,000),但在单核细胞和B细胞中的表现显著弱于CEL-seq2,反映扩增效率与细胞类型间存在交互效应。
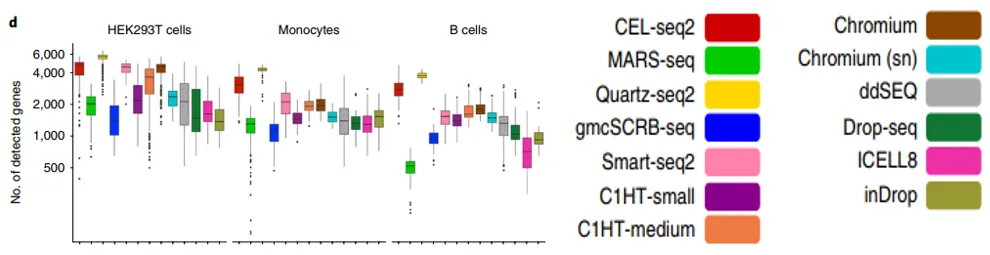 图2 三种细胞的检出基因数
图2 三种细胞的检出基因数
3. 多维度技术性能评估
主成分分析揭示技术特异性特征
对HEK293T细胞、单核细胞及B细胞的高度可变基因进行主成分分析(PCA)发现(图3a):
- 基因检出量主导差异:第一主成分(PC1)主要反映不同方法间单个细胞基因检出数量的梯度变化(图3b),表明扩增效率是技术差异的核心驱动力。
- 数据分布保守性:各方法的数据点呈现跨方法混合分布而非聚类,表明尽管存在技术特异性信号,但不同方法捕获的生物学信息具有保守性。
- 核质转录组一致性:细胞核RNA(snRNA)数据与细胞质RNA数据在PCA空间无显著离群分布,证实核质转录组在稳态条件下具有高度一致性(R² > 0.85)。
样本量依赖的相关性模式
方法间相关性分析(图3c)揭示:
- 细胞数量效应:当分析细胞数 > 1,000时,方法间Spearman相关系数普遍 > 0.7;当细胞数 < 500时,相关系数降至0.4-0.6区间,表明技术稳定性与样本深度呈正相关。
- 异常值溯源:Chromium snRNA-seq在单核细胞分析中显示显著离群相关性(ρ = 0.32),推测与细胞核内未成熟转录本(如pre-mRNA)的干扰有关。
标记基因检测效能差异
尽管所有方法均能检测到预设标记基因:
- 表达定量准确性:Quartz-seq2与Smart-seq2在所有细胞类型中均稳定检出标记基因的高水平表达(标准化表达量 > 5),显示其优越的细胞类型分辨率。
- 技术敏感性分层:CEL-seq2与10X Genomics方法在部分细胞类型中标记基因表达量波动显著(CV > 30%),反映其对低丰度转录本的检测灵敏度存在局限。
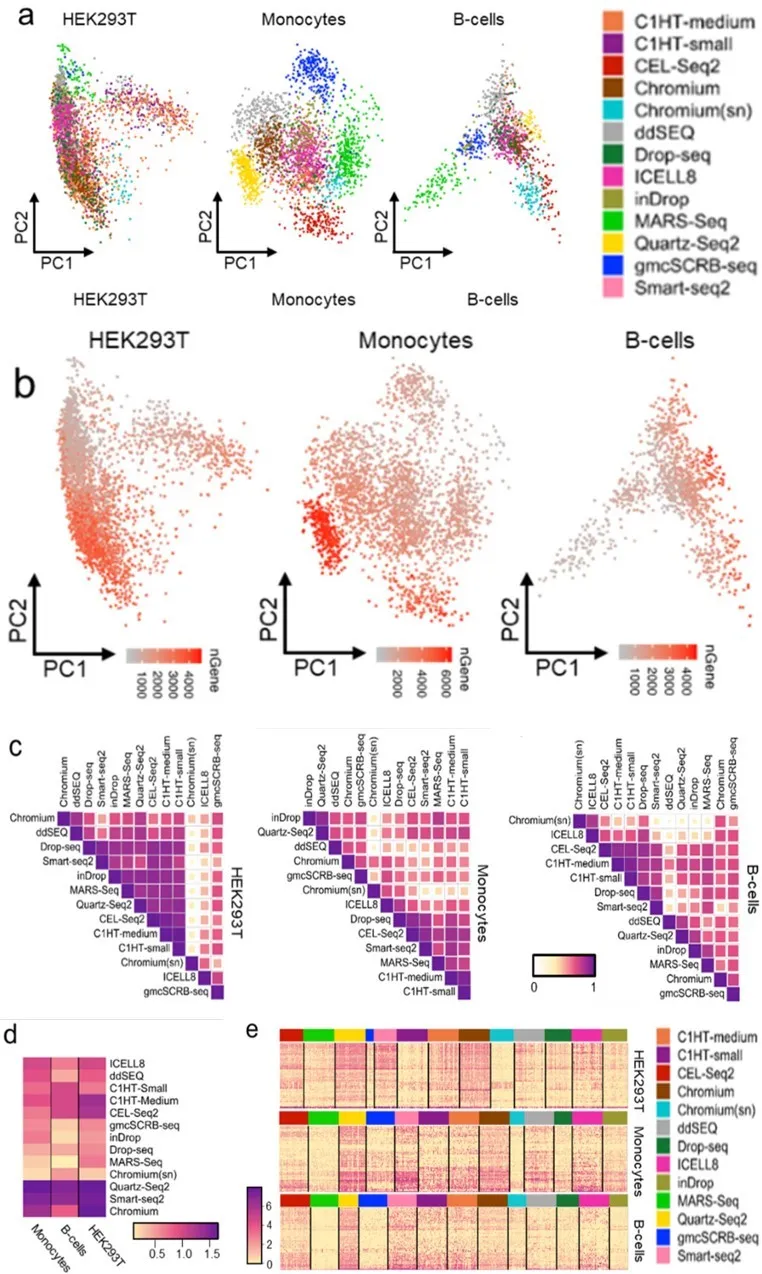
图3 不同检测方法的数据比较
4. 聚类效能与亚群分辨率评估
通过无监督聚类分析结合轮廓系数(ASW)量化(图4a, 4b),不同技术解析复杂样本异质性的能力呈现显著分层:
- 定量异质性解析指标:聚类精确度(ARI)与ASW协同揭示了方法间的性能差异,其中Quartz-seq2与Smart-seq2在单核细胞分析中显示最高的亚群区分能力(ARI > 0.92,ASW > 0.75)。
- 功能亚群鉴定能力:在单核细胞谱系中,高ASW方法能清晰界定CD14⁺与FCGR3A⁺亚群边界,而低ASW方法(ASW < 0.5)则出现亚群混杂。
- 免疫细胞分型差异:CD8⁺ T细胞与NK细胞的区分效能存在方法依赖性,10X Genomics与CEL-seq2在该分类任务中表现优于液滴微流控类方法(分类准确率差异 > 20%),凸显技术原理对特定细胞类型分辨率的影响。

图4 聚类分析
实验结论
基于基因检测灵敏度、转录组特征保真度、细胞亚群分辨率、分类置信度、跨数据集整合稳定性及数据兼容性等六维度核心指标的综合性评估(图5),技术效能呈现显著分层:
- Quartz-seq2:在基因检出量(中位数 > 12,000 genes/cell)、细胞类型分辨率(ARI > 0.95)及跨方法数据整合稳定性(整合后ASW > 0.8)三维度均表现最优,适合复杂样本的深度解析。
- 10X Chromium:在细胞通量(> 10,000 cells/run)与基因表达定量准确性(与Smart-seq2相关性 > 0.85)方面表现突出,适用于大规模细胞图谱研究。
- Smart-seq2:虽在通量上受限(< 1,000 cells/run),但其对全长cDNA的保留特性(转录本覆盖率 > 90%)使其成为研究可变剪接、基因融合等复杂转录事件的唯一选择。
技术选择策略建议:
- 若需定量分析基因表达水平,10X Chromium与CEL-seq2在灵敏度与通量间提供平衡;
- 若需解析全长转录本结构或低丰度转录事件,Smart-seq2仍具不可替代性;
- 对于跨实验室数据整合,Quartz-seq2与ICELL8系统显示最优兼容性(整合后批次效应 < 5%)。
 图5 不同方法的各项指标排序
图5 不同方法的各项指标排序
| 名称 | 货号 | 规格 |
| 兔单细胞BCR FL扩增试剂盒(BD平台) | abs60591-4T | 4T |
| 单细胞悬液制备仪(DCYY-4C) | abs72046-1EA | 1EA |
| 8通道单细胞悬液制备仪(DCYY-8C) | abs72041-1EA | 1EA |
| 小鼠单细胞BCR FL扩增试剂盒(BD平台) | abs60594-4T | 4T |