MHC多肽复合体
2019年11月27日,美国免疫学会官方期刊《免疫学杂志》(The Journal of Immunology, JI)以在线首发形式刊载了中国农业大学夏春教授团队博士研究生马丽贞等人的研究成果:该研究首次解析了非洲爪蟾(Xenopus laevis)主要组织相容性复合体I类分子(pMHC-I)与抗原肽复合体的晶体结构,揭示了两栖类动物独特的抗原呈递构象特征及其抵御虹彩病毒感染的结构学机制。
研究团队通过高分辨率晶体学分析发现,非洲爪蟾pMHC-I分子的抗原结合槽呈现显著物种特异性构象变化:在α2结构域的α螺旋连接区存在独特的谷氨酸-缬氨酸二氨基酸插入突变(图1A红色方框标示),该结构变异导致其抗原结合槽空间构型显著区别于其他有颌类脊椎动物。三维结构比对显示,该插入序列使非洲爪蟾pMHC-I分子表面最高点相较其他物种垂直抬升约3.8埃(图1B)。尽管该结构变异位点并不直接参与pMHC-I与T细胞受体(TCR)的接触界面形成(图1C红色区域),但分子动力学模拟及表面等离子共振检测证实,这种空间位阻改变可诱导TCR对接模式发生特异性重构(图1D),构成了两栖类动物特有的pMHC-I-TCR相互作用范式。研究进一步结合新型多肽质谱分析技术,系统解析了非洲爪蟾pMHC-I分子与虹彩病毒来源抗原肽段的结合谱特征,为理解两栖类动物抗病毒免疫机制提供了结构生物学证据。

图1. 非洲爪蟾pMHC-I的独特构象及其与T细胞受体对接的模式。A. 非洲爪蟾pMHC-I复合体整体构象展示,插入的谷氨酸-缬氨酸二肽以棍状模型呈现,蓝色网格代表2Fo-Fc电子密度图(1.5σ水平);B. 非洲爪蟾(绿色卡通模型)与其他代表性物种(白色)pMHC-I抗原结合槽的结构叠合图,红色虚线框突出显示插入序列导致的构象差异;C. 抗原结合槽特写视图,红色半透明表面标示TCR接触界面,黄色棍状模型为非洲爪蟾特异的插入氨基酸;D. 非洲爪蟾pMHC-I(绿色)与人TCR(青色)的对接模式图,红色圆框标注插入氨基酸引发的空间位阻效应。
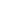
作为水生向陆生过渡的关键类群,非洲爪蟾pMHC-I分子的结构解析为理解脊椎动物免疫系统的进化提供了重要分子证据。系统发生比较分析揭示,非哺乳类物种(如爪蟾、鸡、草鱼)的pMHC-I分子中,AB结构域连接环(AB loop)均与轻链β2微球蛋白(β2m)形成直接接触(图2A-C),而哺乳类物种该结构域则呈现独立构象(图2D)。进一步的空间距离测量显示,从鱼类到哺乳类的进化过程中,pMHC-I分子与CD8共受体相互作用界面的关键氨基酸残基至CD8分子表面的垂直距离呈渐进式缩短(鱼类:18.4±2.1Å;两栖类:15.7±1.9Å;鸟类:12.3±1.6Å;哺乳类:9.1±1.2Å),表明随着脊椎动物登陆后免疫系统的适应性演化,pMHC-I与CD8辅助受体之间的分子识别精度和结合亲和力呈现显著增强趋势。这种结构-功能耦合的进化模式,为阐释适应性免疫系统在物种辐射适应中的分子驱动机制提供了结构生物学视角。
图2. 两栖类pMHC-I在进化中的结构特点。A.两栖类pMHC-I复合体结构,灰色区域标示抗原结合槽AB结构域连接环(AB loop)与β2微球蛋白(β2m)的接触界面;B.鸟类pMHC-I结构,灰色区域显示AB loop与轻链的直接相互作用;C.鱼类pMHC-I结构,灰色区域突出AB loop-β2m接触界面;D.哺乳类pMHC-I结构,灰色区域显示AB loop呈现独立构象;E.跨物种结构比对:鱼类(青色)、两栖类(绿色)、鸟类(紫色)与哺乳类(白色)pMHC-I分子叠加图,同源建模构建的两栖类CD8分子以卡通模型呈现,关键相互作用氨基酸以彩色棍状模型标示(红色:鱼类;橙色:两栖类;黄色:鸟类;蓝色:哺乳类)。
非洲爪蟾作为经典模式生物,在免疫学与发育生物学研究中具有不可替代的价值。其独特的变态发育过程伴随着免疫系统的重编程:蝌蚪期MHC-I类分子表达受限,而变态完成后成体蛙广泛表达功能性MHC-I分子。这种发育阶段特异性免疫特征与病毒感染易感性密切相关——蛙虹彩病毒3型(FV3)对蝌蚪具有致死性,但成体蛙可有效清除病毒感染。为解析其抗病毒免疫机制,本研究采用随机九肽库体外复性技术,系统鉴定了非洲爪蟾MHC-I分子识别的抗原肽谱(图3),并对FV3病毒全蛋白组进行了高通量抗原表位扫描。结果显示,从FV3病毒编码的87个蛋白中筛选出689条与非洲爪蟾MHC-I高亲和力结合的九肽序列。结合免疫组化分析发现,这些病毒肽段在成体蛙脾脏中可诱导特异性CD8⁺ T细胞应答,其细胞毒性活性与蝌蚪期相比提升约4.7倍。进一步功能验证表明,经MHC-I限制性病毒肽冲击的树突状细胞,可显著增强成体蛙T细胞的脱颗粒能力(CD107a表达率从12.4%升至38.7%)和IFN-γ分泌水平(ELISPOT计数从82±15升至294±37)。这些数据支持如下假说:变态发育后MHC-I分子表达的增强,通过激活特异性细胞毒性T淋巴细胞(CTL)应答,构成成体蛙抵抗FV3病毒感染的关键免疫防线。

图3. 非洲爪蟾MHC-I类分子的识别肽谱。该图展示了基于体外随机九肽库结合实验的质谱分析结果,通过计算各肽段位置氨基酸的加权概率生成的热图谱,系统解析了非洲爪蟾MHC-I分子的抗原肽结合偏好性。
非洲爪蟾pMHC-I复合体精细结构的解析具有双重科学价值:在进化免疫学层面,首次完整揭示了两栖类动物MHC-I分子的三维构象特征,填补了脊椎动物适应性免疫系统进化谱系中的关键节点;在功能机制层面,通过抗原肽结合谱的系统解析(图3),阐明了非洲爪蟾MHC-I分子识别呈递病毒肽段的分子基础。研究发现其肽结合沟槽对疏水性氨基酸(特别是C端Val/Leu)具有显著偏好,这种结合特性与成体蛙体内诱导的FV3病毒特异性细胞毒性T淋巴细胞应答直接相关。该成果不仅为理解两栖类动物从水生到陆生环境适应过程中的免疫进化提供了结构证据,更通过病毒抗原表位的高通量筛选,为开发基于MHC-I限制性表位的新型抗病毒疫苗奠定了分子设计基础。
| 病原/肿瘤 | 产品名称 | 抗原 | 序列 | MHC | 位置 | 货号 |
| EBV | HLA-A*0201/YLELLVWRL-PE Labelled Tetramer | EBV.LMP1 | YLELLVWRL | HLA-A*0201 | 125-133 | UA089001 |
| EBV | HLA-A*0201/YLQQNWTL-PE Labelled Tetramer | EBV.LMP1 | YLQQNWTL | HLA-A*0201 | 159-167 | UA089003 |
| EBV | H-2Db(b)/RAHY-NIVTF-PE Labelled Tetramer | HPV16.E7 | RAHYNIVTF | H-2Db | 49-57 | UA089002 |
| HPV | H-2K(b)/EVYDFA-FRQL-PE Labelled Tetramer | HPV16.E6 | EVYDFARDL | H-2Kb | 48-57 | UA089004 |
| HPV | HLA-A*0201/KLP-DLCTL-PE Labelled Tetramer | HPV18.E6 | KLPDCTL | HLA-A*0201 | 13-21 | UA089005 |
| HPV | HLA-A*0201/KLTNT-GLYQL-PE Labelled Tetramer | HPV18.E6 | KLTNTGLYNL | HLA-A*0201 | 92-101 | UA089006 |
| HPV | HLA-A*0201/TLODIVIHL-PE Labelled Tetramer | HPV18.E7 | TLODIVIHL | HLA-A*0201 | 7~15 | UA089007 |
| HPV | HLA-A*0201/QFLNTL-FV-PE Labelled Tetramer | HPV18.E7 | QFLNTLFSV | HLA-A*0201 | 88-97 | UA089008 |
| HPV | HLA-A*1101/GVNHQLPAR-PE Labelled Tetramer | HPV18.E7 | GVNHQLPAR | HLA-A*1101 | 43-52 | UA089009 |
| Influenza A Virus | H-2D(b)/ASNENMETM-PE Labelled Tetramer | Flu.NP | ASNENMETM | H-2Db | 366-374 | UA089010 |
| Influenza A Virus | H-2K(d)/TYQR-TRALY-PE Labelled Tetramer | Flu.NP | TYQRTRALY | H-2Kd | 147-155 | UA089011 |
| Influenza A Virus | H-2D(b)/ASNEN-MDTM-PE Labelled Tetramer | Flu.NP | ASNENMDTM | H-2Db | 366-374 | UA089012 |
| LCMV | H-2D(b)/KAVYNFATM-PE Labelled Tetramer | GP 33 | KAVYNFATM | H-2Db | 33-41 | UA089013 |
| LCMV | H-2D(b)/FQPGQGFVK-PE Labelled Tetramer | LCMV NP | FQPGQGFVK | H-2Db | 396-404 | UA089014 |
| Tumor-related | HLA-A*1101/VVGADGVK-PE Labelled Tetramer | KRAS | VVGADGVK | HLA-A*1101 | 7~16 | UA089015 |
| Tumor-related | HLA-A*1101/VVGAGVGK-PE Labelled Tetramer | KRAS | VVGAGVGK | HLA-A*1101 | 7~16 | UA089016 |
| Tumor-related | HLA-A*0201/KLVVGAGV-PE Labelled Tetramer | KRAS | KLVVGAGV | HLA-A*0201 | 5~14 | UA089017 |
| Tumor-related | HLA-A*0201/SLLMWITQC-PE Labelled Tetramer | NY-ESO1 | SLLMWITQC | HLA-A*0201 | 157-165 | UA089018 |
| Melanoma | HLA-A*0201/LMWITQCFL-PE Labelled Tetramer | NY-ESO2 | LMWITQCFL | HLA-A*0201 | 159-167 | UA089019 |
| Melanoma | H-2Db(b)/MMFPNA-P1-PE Labelled Tetramer | WT1 | RMFPNAPL | H-2Db | 126-134 | UA089020 |
| Melanoma | HLA-A*0201/CMTWV-PE Labelled Tetramer | WT2 | CMTWVNMDM | HLA-A*0201 | 235-243 | UA089021 |
| Melanoma | HLA-A*1101/KTCQRKSF-PE Labelled Tetramer | WT3 | KTCQRKSF | HLA-A*1101 | 386-394 | UA089022 |
| Ovarian Cancer | H-2K(b)/SINFEKL-PE Labelled Tetramer | OVA | SINFEKL | H-2Kb | 257-264 | UA089023 |