MHC I多聚体技术
您是否了解MHC I 多聚体的类型有哪些?Tetramers、Pentamers、Streptamers之间有哪些区别?
在IBA Lifesciences发布的《揭秘MHC I多聚体的世界》一文中,对MHC多聚体类型、差异、特性、以及发展前景进行了归纳与总结,全文免费下载地址见本篇文末。
MHC多聚体——免疫学研究的革命性工具
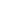
细胞毒性T细胞(Cytotoxic T lymphocytes,CTL)作为免疫应答的核心效应细胞,通过其表面T细胞受体(TCR)特异性识别抗原,执行关键的免疫监视功能。在抗原识别过程中,CTL的CD8共受体与TCR协同作用,精准识别由MHC I类分子呈递的抗原肽。
MHC I类分子广泛表达于所有有核细胞表面,其核心功能是结合并展示细胞内源性蛋白降解产生的肽段,包括病毒蛋白、肿瘤相关抗原等。即使这些肽段源自自身蛋白,一旦被MHC I类分子识别为异常抗原,具有对应TCR的CTL即可启动细胞毒作用,精确清除受感染或发生癌变的细胞,从而有效遏制感染扩散和肿瘤进展。
每个T细胞受体(TCR)通过特异性识别独特的肽-MHC(pMHC)复合物执行免疫监视功能,为应对环境中海量的抗原变异,免疫系统需维持庞大的TCR多样性库。这种多样性主要通过基因层面的组合机制实现:在T细胞发育过程中,V(D)J基因片段重排、连接区域核苷酸的随机切除或添加、以及αβ异源二聚体链的随机配对,共同构建了TCR的多样性基础。理论计算表明,上述机制可产生高达10^20种独特的TCR分子,为机体提供了近乎无限的抗原识别潜能。
全面解析免疫应答机制并深入理解其生物学基础,需对参与免疫识别的T细胞及其T细胞受体(TCR)展开系统性研究。这一研究领域不仅属于基础科学范畴,更直接关联疫苗研发等应用领域。例如,在病毒感染周期或疫苗接种后,对T细胞亚群的动态分析可揭示免疫应答的质量、持续时间和保护性特征,为评估免疫干预效果提供关键指标。
为突破抗原特异性T细胞的研究瓶颈,Altman教授课题组于1996年开创性地提出将肽-MHC I类分子(pMHC I)复合物作为分子探针的技术方案。其核心创新在于:通过化学交联技术将多个pMHC I单体组装于荧光标记的骨架结构,形成多价复合物。这种设计显著增强了探针与TCR的结合亲和力,成功实现对HIV特异性T细胞的直接染色与流式分析。该研究奠定了MHC多聚体技术的理论基础,证实低亲和力pMHC I单体通过空间聚集效应可实现功能强化,从而稳定结合靶细胞表面TCR。历经近三十年发展,MHC多聚体技术已成为抗原特异性T细胞研究的标准工具,持续推动着适应性免疫应答的机制解析与临床转化研究。
MHC I 多聚体有哪些类型?
四聚体(TETRAMERS)
1996年开发的首个MHC I类多聚体技术被称为MHC I四聚体,其核心结构由4个肽-MHC I类分子(pMHC I)复合体共价结合至抗生物素蛋白(Avidin)骨架构成。抗生物素蛋白是一种源自卵清的四聚体糖蛋白,每个亚基含有一个高亲和力生物素结合位点,可同时结合4个生物素标记的pMHC I单体。作为替代方案,链霉亲和素(Streptavidin)——一种来源于链霉菌的非糖基化抗生物素蛋白类似物——因其更低的非特异性结合背景,逐渐成为四聚体构建的优选骨架。通过将荧光染料共价偶联至骨架蛋白,四聚体复合物可实现对抗原特异性T细胞的直接流式染色。四聚体技术的诞生奠定了MHC多聚体工具的开发基础,并持续推动着抗原特异性T细胞检测技术的优化。除经典四聚体外,目前商业化的MHC I类多聚体产品还包括五聚体(Pentamers)、链霉亲和素多聚体(Streptamers)以及右旋糖酐多聚体(Dextramers)等,各类型多聚体在骨架设计、结合动力学及实验应用场景方面展现出差异化优势。此外,新型多聚体技术如NTAmers等亦在不断发展,进一步丰富了MHC多聚体的技术谱系。
五聚体(PENTAMERS)
MHC I类五聚体技术于2000年问世,其创新结构由5个pMHC I复合物共价组装于自组装型卷曲螺旋骨架构成。该卷曲螺旋结构域源自软骨基质蛋白(Cartilage Oligomeric Matrix Protein, COMP)的天然多聚化模块,通过基因工程改造实现5个pMHC I分子的定向排列,确保所有抗原结合位点呈空间同向性分布。这种设计显著提升了表位密度,使五聚体与TCR的亲和效能较传统四聚体提升约10倍。相较于四聚体技术,五聚体可同时偶联5个荧光基团或生物素标记,其单位面积荧光信号强度提高40%-60%,在低频抗原特异性T细胞检测中表现出更高的标记效率和检测灵敏度,尤其适用于肿瘤新抗原或病毒变异株特异性T细胞的稀有细胞群分析。
STREPTAMERS
MHC I类Streptamer技术基于Strep-tag®系统开发,于2002年首次实现商业化应用。该技术通过基因融合策略,将pMHC I分子的C端与双链霉亲和素标签(Twin-Strep-tag®)偶联,使其能够以高亲和力(Kd≈10⁻⁸ M)结合链霉亲和素突变体Strep-Tactin®。其核心创新在于引入竞争性解离机制:添加游离生物素后,高亲和力生物素通过竞争性置换实现pMHC I分子的可控解离,使Streptamer复合物成为首个可逆性MHC I类多聚体工具。这一特性使其在T细胞分选、体外功能实验及体内回输治疗研究中具有独特优势,可有效避免传统多聚体染色导致的TCR信号持续激活。
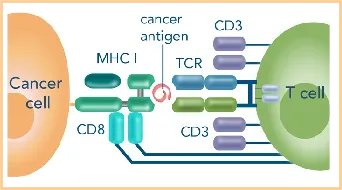
DEXTRAMERS
MHC I类Dextramer技术是继Streptamer之后开发的第二代多聚体系统,其命名源自葡聚糖(Dextran)骨架结构。该技术通过化学交联将多个pMHC I分子共价偶联至线性葡聚糖纤维,形成高密度表位展示平台。葡聚糖骨架的分支结构使每个复合物可负载多达50个pMHC I分子,显著提升了表位呈现效率。实验数据显示,Dextramer技术对低亲和力TCR(解离速率常数Kd>10⁻⁵ M)的检测灵敏度较传统四聚体提升10-100倍,成功实现了对肿瘤新抗原特异性T细胞等稀有细胞群的直接检测,填补了传统方法在低亲和力表位研究中的技术空白。
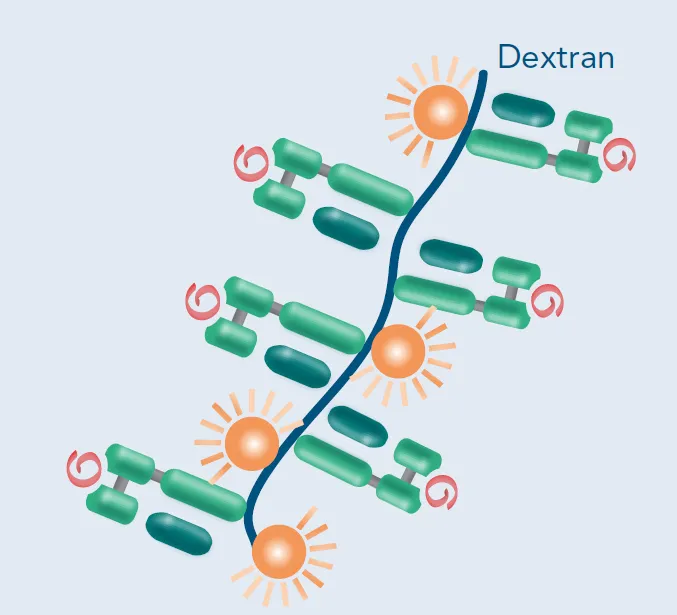
NTAMERS
NTAmer技术代表第三代可逆性MHC I类多聚体发展方向,其命名源于镍-氨三乙酸(Ni²+-NTA)螯合系统。该技术通过基因工程在pMHC I复合物中引入6×组氨酸标签(His-tag),利用Ni²+-NTA与组氨酸的金属螯合作用实现多聚化。其解聚机制与Streptamer技术类似:添加咪唑竞争性结合Ni²+后,金属螯合键断裂导致多聚体解离。尽管目前尚未有商业化NTAmer产品,但前期研究表明,该系统在TCR-pMHC相互作用动力学研究及长时间活细胞成像中具有潜在应用价值,其解离速率可调特性为解析TCR信号转导的时空调控提供了新型工具。
可逆性——MHC多聚体的重要特性
通过可逆性这一核心特征,可清晰区分不同MHC I类多聚体技术体系。传统MHC多聚体(如四聚体、五聚体)通过共价交联或高亲和力非共价作用实现pMHC I分子的多聚化,显著提升表位密度与TCR结合强度。然而,这种不可逆结合模式可能导致染色复合物内化、T细胞功能抑制或凋亡等负面效应,并限制TCR-配体解离动力学研究。
技术原理对比
经典四聚体依赖抗生物素蛋白-生物素系统(Kd≈10⁻¹⁵ M),形成超稳定复合物,但无法实现可控解离;五聚体与葡聚糖多聚体(Dextramer)虽通过表位密度优化提升检测灵敏度,仍维持不可逆结合特性。相较之下,MHC I类Streptamer技术采用Twin-Strep-tag®与链霉亲和素突变体Strep-Tactin®的特异性结合(Kd≈10⁻⁸ M),通过添加游离生物素竞争性解离,实现pMHC I分子的可控脱落。NTAmer技术则基于Ni²+-NTA螯合系统,利用咪唑介导的His-tag解离,提供另一种可逆化解决方案。
MHC I STREPTAMER 细胞染色实验流程
MHC I类Streptamer试剂为抗原特异性T细胞检测与分选提供了革新性工具。其双色荧光标记系统(Streptamer复合物+抗Strep-Tactin®荧光抗体)可实现低至0.001%的稀有细胞群精准鉴定,信噪比较传统四聚体提升5-8倍。
该技术核心优势在于:
- 无标记分选:染色后通过生物素洗脱可获得完全去标记的活细胞,避免荧光残留干扰下游功能实验;
- 功能完整性保留:解离后的T细胞维持完整增殖能力与效应功能,可直接用于体外扩增、杀伤实验或过继回输研究;
- 动态分析兼容性:支持实时监测TCR-pMHC相互作用动力学,为免疫突触形成机制研究提供技术平台。
临床前研究显示,Streptamer分选的肿瘤浸润淋巴细胞(TIL)在体外扩增后,其抗原特异性保持率较传统方法提高3倍,为个性化细胞治疗产品开发奠定了方法学基础。
相关商品推荐
| 病原/肿瘤 | 产品名称 | 抗原 | 序列 | MHC | 位置 | 货号 |
| EBV | HLA-A*0201/YLELLVWRL-PE Labelled Tetramer | EBV.LMP1 | YLELLVWRL | HLA-A*0201 | 125-133 | UA089001 |
| EBV | HLA-A*0201/YLQQNWTL-PE Labelled Tetramer | EBV.LMP1 | YLQQNWTL | HLA-A*0201 | 159-167 | UA089003 |
| EBV | H-2Db(b)/RAHY-NIVTF-PE Labelled Tetramer | HPV16.E7 | RAHYNIVTF | H-2Db | 49-57 | UA089002 |
| HPV | H-2K(b)/EVYDFA-FRQL-PE Labelled Tetramer | HPV16.E6 | EVYDFARDL | H-2Kb | 48-57 | UA089004 |
| HPV | HLA-A*0201/KLP-DLCTL-PE Labelled Tetramer | HPV18.E6 | KLPDCTL | HLA-A*0201 | 13-21 | UA089005 |
| HPV | HLA-A*0201/KLTNT-GLYQL-PE Labelled Tetramer | HPV18.E6 | KLTNTGLYNL | HLA-A*0201 | 92-101 | UA089006 |
| HPV | HLA-A*0201/TLODIVIHL-PE Labelled Tetramer | HPV18.E7 | TLODIVIHL | HLA-A*0201 | 7~15 | UA089007 |
| HPV | HLA-A*0201/QFLNTL-FV-PE Labelled Tetramer | HPV18.E7 | QFLNTLFSV | HLA-A*0201 | 88-97 | UA089008 |
| HPV | HLA-A*1101/GVNHQLPAR-PE Labelled Tetramer | HPV18.E7 | GVNHQLPAR | HLA-A*1101 | 43-52 | UA089009 |
| Influenza A Virus | H-2D(b)/ASNENMETM-PE Labelled Tetramer | Flu.NP | ASNENMETM | H-2Db | 366-374 | UA089010 |
| Influenza A Virus | H-2K(d)/TYQR-TRALY-PE Labelled Tetramer | Flu.NP | TYQRTRALY | H-2Kd | 147-155 | UA089011 |
| Influenza A Virus | H-2D(b)/ASNEN-MDTM-PE Labelled Tetramer | Flu.NP | ASNENMDTM | H-2Db | 366-374 | UA089012 |
| LCMV | H-2D(b)/KAVYNFATM-PE Labelled Tetramer | GP 33 | KAVYNFATM | H-2Db | 33-41 | UA089013 |
| LCMV | H-2D(b)/FQPGQGFVK-PE Labelled Tetramer | LCMV NP | FQPGQGFVK | H-2Db | 396-404 | UA089014 |
| Tumor-related | HLA-A*1101/VVGADGVK-PE Labelled Tetramer | KRAS | VVGADGVK | HLA-A*1101 | 7~16 | UA089015 |
| Tumor-related | HLA-A*1101/VVGAGVGK-PE Labelled Tetramer | KRAS | VVGAGVGK | HLA-A*1101 | 7~16 | UA089016 |
| Tumor-related | HLA-A*0201/KLVVGAGV-PE Labelled Tetramer | KRAS | KLVVGAGV | HLA-A*0201 | 5~14 | UA089017 |
| Tumor-related | HLA-A*0201/SLLMWITQC-PE Labelled Tetramer | NY-ESO1 | SLLMWITQC | HLA-A*0201 | 157-165 | UA089018 |
| Melanoma | HLA-A*0201/LMWITQCFL-PE Labelled Tetramer | NY-ESO2 | LMWITQCFL | HLA-A*0201 | 159-167 | UA089019 |
| Melanoma | H-2Db(b)/MMFPNA-P1-PE Labelled Tetramer | WT1 | RMFPNAPL | H-2Db | 126-134 | UA089020 |
| Melanoma | HLA-A*0201/CMTWV-PE Labelled Tetramer | WT2 | CMTWVNMDM | HLA-A*0201 | 235-243 | UA089021 |
| Melanoma | HLA-A*1101/KTCQRKSF-PE Labelled Tetramer | WT3 | KTCQRKSF | HLA-A*1101 | 386-394 | UA089022 |
| Ovarian Cancer | H-2K(b)/SINFEKL-PE Labelled Tetramer | OVA | SINFEKL | H-2Kb | 257-264 | UA089023 |