该操作步骤描述了在 Illumina 平台上进行测序的文库构建。
如下操作步骤以产品货号180419的步骤为例,产品列表详见右下方推荐产品
实验步骤
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
-
根据表格1,将组分添加到含有纯化 PCR 产物的 PCR 管或板中。
注意:使用条形码适配器时,一次打开一个适配器管并在移取不同的条形码适配器之间更换手套,以避免交叉污染。
表 1. PCR 产物接头连接试剂零件 体积/反应(微升) 从基因组或多重 PCR 产物中纯化的 PCR 扩增子 视情况而定; 10–100 纳克* 4x 1-Step Amplicon Library Buffer 12.5 QIAseq CDI 或 UDI Y-Adapter Plate (96) 4 或者 GeneRead 12-plex 接头(货号180985或 180986) 2 1-Step Amplicon Enzyme Mix 2 无DNA酶水 视情况而定 全部的 50 * 我们建议通过微流体或毛细管电泳平台对 PCR 产物进行定量。如果使用其他供应商的适配器,请将适配器添加到 1 µM 最终浓度,并按照供应商的说明进行操作。
-
混合组分
通过上下移液几次来混合组分。
-
将热循环仪编程为在 25°C 下孵育 30 分钟
将热循环仪编程为在 25°C 下孵育 30 分钟。视情况其他反应可以在室温下孵育。
重要提示:请勿使用带加热盖的热循环仪。 -
将反应物置于冰上并使用 Agencourt AMPure XP 珠进行纯化
反应完成后,将反应物置于冰上并使用 Agencourt AMPure XP 珠进行纯化。
-
为每个连接反应准备 1.5 ml LoBind 管并标记试管或准备 96 孔板
为每个连接反应准备 1.5 ml LoBind 管并标记试管或准备 96 孔板(具体选择哪个取决于磁架的可用性和个人喜好)。
-
转移到准备好的 1.5 ml LoBind 管或 96 孔板中,并加入 50 µl 无核酸酶水
将步骤 4 中的 50 µl 连接反应液转移到准备好的 1.5 ml LoBind 管或 96 孔板中,并加入 50 µl 无核酸酶水。
-
加入Agencourt AMPure XP 微珠
对于长度不超过 350 bp 的 PCR 产物:
向每个连接反应中加入 40 µl(0.4 倍体积)Agencourt AMPure XP 微珠,通过移液器充分混合,然后继续执行步骤 8。
对于大小为 350–1000 bp 的 PCR 产物:
在每个连接反应中加入 60 µl(0.6 倍体积)Agencourt AMPure XP 微珠,通过移液器充分混合,然后继续执行步骤 12(跳过步骤 8-11)。 -
室温下孵育混合物 5 分钟
在室温下孵育混合物 5 分钟。将磁珠放在磁性支架(例如 DynaMag)上 5 分钟。
-
为每个连接反应准备一个新的 LoBind 管或一个新的 96 孔板。
为每个连接反应准备一个新的 LoBind 管或一个新的 96 孔板。
-
将 133 µl 上清液转移到新管
小心地将 133 µl 上清液转移到新管中,不要干扰磁珠。这将留下大约 7 µl 上清液。丢弃含有不需要的大 DNA 片段的珠子。大DNA 片段是通过接头与非特异性多重 PCR 产物的连接产生的。
注意:不要丢弃上清液。 -
将 40 µl 重悬的 Agencourt AMPure XP Beads 浆液添加到上清液中
将 40 µl 重悬的 Agencourt AMPure XP Beads 浆液添加到上清液中,并通过移液器充分混合。
-
在室温下孵育混合物 5 分钟
在室温下孵育混合物 5 分钟。
-
脉冲旋转管或板
脉冲旋转管或板。将磁珠放在磁性支架上(例如 DynaMag)5 分钟,然后小心地取出并丢弃上清液。小心不要丢弃珠子,因为它们包含文库。
注意:不要丢弃珠子。 -
加入 200 µl 80% 乙醇清洗磁珠
加入 200 µl 80% 乙醇清洗磁珠。将磁珠放在磁力架上,弃去上清液。重复洗涤一次,总共进行 2 次乙醇洗涤。
-
尽量在不干扰珠子的情况下去除残留的乙醇
尽量在不干扰珠子的情况下去除残留的乙醇。在磁力架上孵育珠子 5-10 分钟或直到珠子干燥。珠子过度干燥可能会导致 DNA 回收率降低。然后从磁性支架上取下。
-
通过重悬于 26 µl 无核酸酶水中进行洗脱
通过重悬于 26 µl 无核酸酶水中进行洗脱。通过移液充分混合并在室温下孵育管或板 2 分钟以从珠子中洗脱 DNA。
-
将管或板放回磁架上以使珠子沉淀
将管或板放回磁架上以使珠子沉淀。孵育直到液体澄清。
-
文库扩增过程中使用 23.5 µl 洗脱液
在文库扩增过程中使用 23.5 µl 洗脱液,如果输入的扩增子数量足够,则直接进行定量和测序。
-
根据以下内容对带有加热盖的热循环仪编程表 2
表 2. DNA 文库扩增的循环条件
时间 温度 循环次数 初始变性 2 分钟 98°C 1 退火 20 s 98°C 4–10* 30 s 60°C 30 s 72°C 最终延期 1分钟 72°C 1 ∞ 4°C 存储 * 注:循环数取决于输入扩增子的数量和质量。一般来说,4 个循环足以处理 20-500 ng 的 PCR 输入产物,10 个循环足以处理 1-20 ng 的 PCR 产物输入。如果输入 DNA 足够(>500 ng),则可以省略文库扩增。
-
在 0.2 ml PCR 管或 96 孔 PCR 板中混合表 3 中的成分。
表 3. PCR 扩增反应组分
零件 体积(微升) 高保真 PCR 预混液,2x 25 底漆混合物(每个 10 µM) 1.5 文库 DNA(来自上一步) 23.5 全部 50 -
将 PCR 板转移到热循环仪并启动程序
将 PCR 板转移到热循环仪并启动程序。
-
将 50 µl 重悬的 Agencourt AMPure XP Beads 添加到每个反应 (50 µl) 中
PCR 完成后,将 50 µl 重悬的 Agencourt AMPure XP Beads 添加到每个反应 (50 µl) 中,并上下吸管彻底混合珠子和 PCR 混合物。
-
室温下孵育混合物 5 分钟
在室温下孵育混合物 5 分钟。将磁珠放在磁性支架(例如 DynaMag)上,小心丢弃上清液。
-
加入 200 µl 80% 乙醇清洗磁珠
加入 200 µl 80% 乙醇清洗磁珠。将磁珠放在磁力架上,弃去上清液。重复洗涤一次,总共两次乙醇洗涤。从磁性支架上取下。
-
磁力架上孵育 5-10 分钟
在磁力架上孵育 5-10 分钟,直到珠子变干。珠子过度干燥可能会导致 DNA 回收率降低。从磁性支架上取下。
-
通过重悬于 30 µl 无核酸酶水中进行洗脱
通过重悬于 30 µl 无核酸酶水中进行洗脱。通过移液充分混合。将磁珠放在磁力架上。小心地将 28 µl 上清液转移至干净的 LoBind1.5 ml 试管或 PCR 板。
-
使用毛细管电泳设备或类似方法评估库的质量
使用毛细管电泳设备或类似方法评估库的质量。检查文库片段的预期大小分布,是否缺少接头、扩增引物、接头二聚体或高分子量过度扩增伪影。
注意:库应显示反映输入 PCR 扩增子大小加上 120 bp 的分布。文库长度的增加反映了向 PCR 扩增子添加了测序接头。中值片段大小可用于后续基于 qPCR 的量化方法来计算文库浓度。 -
对文库进行量化
使用 GeneRead Library Quant Kit(货号 180612)或类似方法对文库进行量化。
-
纯化的文库可以安全地储存在 –20°C 的 DNA LoBind 管中
纯化的文库可以安全地储存在 –20°C 的 DNA LoBind 管中,直到准备好进行测序。
-
典型结果
使用高质量、无伪影的 PCR 产品时; QIAseq 1-Step Amplicon 文库通常不含接头二聚体、文库扩增引物、过量接头和高分子量扩增产物。当为输入量选择合适的循环数时,纯化后的文库产量应约为 5 nM,并且体积应足以在大多数 NGS 平台上进行质量控制、文库量化和测序。
在示例实验中,如图图3所示, 参考 DNA 使用 GeneRead DNAseq V2 Human Comprehensive Cancer Panel(06/2015 版)扩增。该面板由 4 个独立的 PCR 反应组成,每个反应包括 1988 对引物。扩增子旨在捕获 160 个癌症相关基因,包括大约 745 kb 的基因组。 PCR 后,汇集产物并分析产量和长度分布(图 3).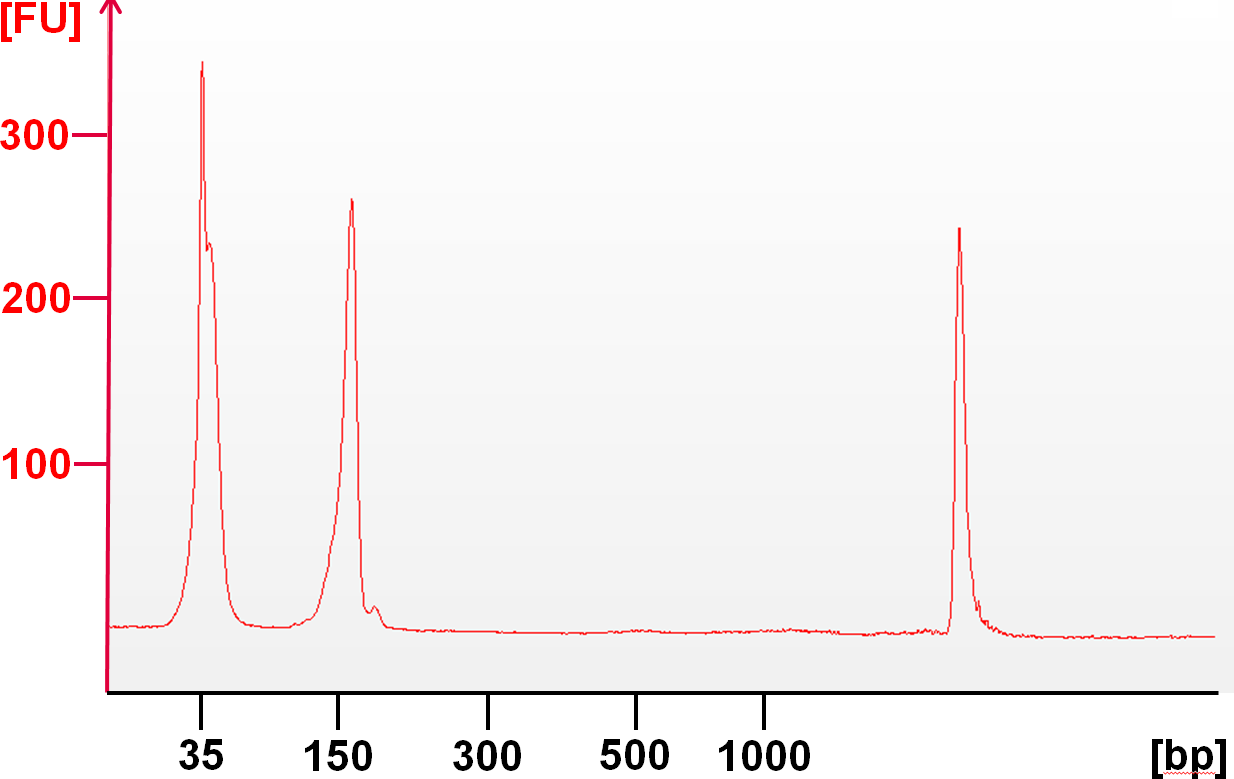
图 3. 混合多重 PCR 扩增子的电泳图谱。在扩增和汇集后,可以看到一个预期大小 (160 bp) 的峰值,它代表这组 PCR 引物的平均扩增子大小。以 35 bp 为中心的第二个大峰由电泳标记、未延伸的引物和延伸的引物二聚体组成。在纯化过程中,这些较短的产物被去除,只留下所需的 PCR 扩增子。△点击放大图片
PCR 产物根据 GeneRead DNAseq Targeted Panels V2 手册使用 Agencourt AMPure XP Beads 进行纯化,并使用 25 ng 使用 QIAseq 1-Step Amplicon Library Kit 生成文库。文库通过 4 个循环的 PCR 扩增,纯化后共产生 28 µl 5 nM 文库(图4, 显示 1:10 稀释)。虽然测序质量、读取长度和获得的读取数量会因测序平台而异,但使用 QIAGEN GeneRead DNAseq Library Quant Array 等文库量化方法可以实现更准确的聚类,优化数据产量和质量。
使用 QIAseq 1-Step Amplicon Library Kit 生成的典型数据包含由接头二聚体产生的最少读数,当与高质量 panel 配对时,通常具有 >95% 的目标读数,在目标扩增子上读数分布均匀。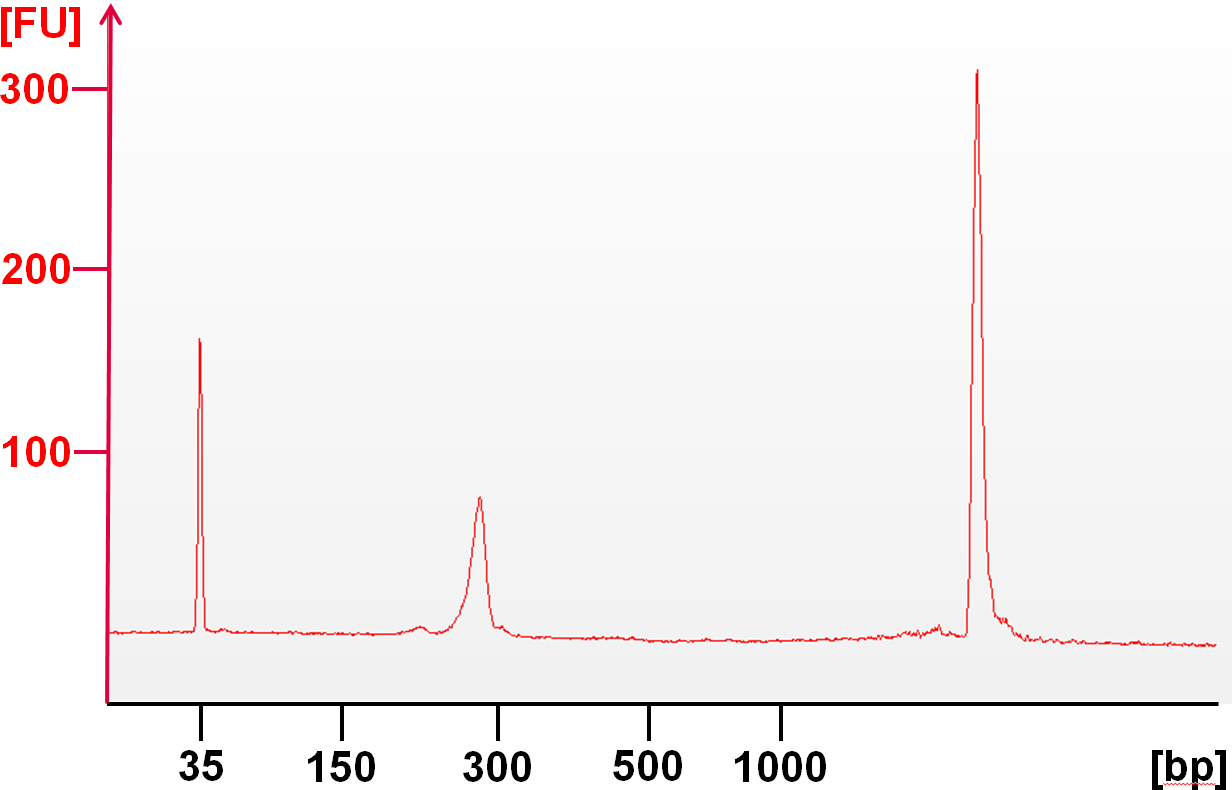
图 4. 使用 QIAseq 1-Step Amplicon Library Kit 制备的纯化文库的电泳图谱。由于接头添加的附加序列,观察到 120 bp 的特征大小变化。典型的文库应不含由过量引物、接头或接头二聚体和高分子量峰组成的较短峰,这些峰可由基因组引入或通过对完整库进行过度扩增△点击放大图片


