如下操作步骤以产品货号333937的步骤为例,产品列表详见右下方推荐产品
实验步骤
- 1
- 2
- 3
- 4
- 5
-
QIAseq 人类外显子组试剂盒
扩增的、索引的全基因组文库的汇集使得能够在单个杂交-捕获反应中处理多个文库。我们建议将最多8个索引文库合并用于一个杂交捕获反应。或者,可以对单个文库进行杂交捕获。要合并的索引库的数量取决于每个库的样本数。确保仅使用PCR扩增文库进行杂交捕获。
开始前要做的事情· 量化扩增的、索引的全基因组文库
· 在冰上解冻人类外显子组探针组、All-4-One Blocking Oligos 和 All-4-One Blocking Solution,然后脉冲涡旋和脉冲自旋。
· 要在文库池干燥后立即进行杂交捕获,请将 Vapor-Lock 试剂平衡至室温,并在池蒸发完成之前将快速杂交溶液加热至 65°C。或者,干燥的文库池可在 –30 至 –15°C 下储存长达 3 天。
程序
1. 对于每个捕获池,根据所需的池大小确定每个库的数量 (ng),参考指南表 7或遵循建议表 8。 确保每个捕获池的总输入不超过 3200 ng。表 7. 为杂交捕获汇集索引库的指南
文库汇集指南 每个捕获池的推荐库数 8 每个捕获池的最大总输入 3200 纳克 每个库的最小输入 200 纳克
表 8. 用于杂交捕获的推荐文库池策略每个池的索引样本数 每个池中每个索引库的数量 1 200–500ng 2 200–500ng 3 200–500ng 4 200–500ng 6 200–500ng 8 200–400ng *每个池的总 DNA 输入量不要超过 3200 ng。
注意:为了在测序期间保持文库的相等表示,对所有文库使用相同的输入量。每个文库使用少于 200 ng 的输入可能会降低复杂性。
2. 计算池所需的每个放大库的体积 (µl),例如,对于 25 ng/µl 的库浓度,需要 16 µl 才能将 400 ng 的索引库添加到捕获池中。
3. 将每个文库的计算体积转移到 1.5 ml LoBind 管中。如果一次进行多个杂交反应,请使用多个试管。
4. 根据表9。表 9. 用探针和封闭试剂补充文库库
零件 体积(微升) 索引库池(来自第 3 步) 多变的 人类外显子组探针组 4 One-4-All 封闭寡核苷酸 8 One-4-All 阻塞解决方案 5
5. 脉冲涡旋补充库池以混合和脉冲旋转管。确保存在最少的气泡。
6. 使用 SpeedVac 系统(或类似的蒸发器设备)完全蒸发补充库池中的所有液体内容。如果需要,通过将温度设置为 60°C 来加速较大体积的蒸发。
7. 当补充库池的蒸发完成时,将干燥的库池储存在冰上,并立即进行步骤 8(杂交捕获操作步骤)。或者,干燥的文库池可在 –30 至 –15°C 下储存长达 3 天。 -
杂交捕获
在杂交步骤中,QIAseq Exome Probes 特异性结合库中存在的目标文库片段。灵活的杂交方案允许将杂交持续时间从 30 分钟调整到 4 小时。孵育 1 小时或更长时间可以提高捕获性能。孵育超过 4 小时或过夜不会显著提高性能,但可以考虑在实验计划中实现最高的灵活性。
开始前要做的事情· 解冻 Vapor-Lock 试剂并平衡至室温。
· 在加热块中将快速杂交溶液加热至 65°C 至少 10 分钟,并确保所有沉淀物都溶解。
· 当进行1小时或更短时间的杂交捕获反应时,开始准备将杂交靶标与链霉亲和素结合珠结合所需的试剂,如下一章开头所述。
程序
8. 将 Vapor-Lock 试剂解冻并平衡至室温。在加热块中将快速杂交溶液加热至 65°C 10 分钟。涡旋并确保所有沉淀物溶解。将快速杂交溶液保持在 65°C 直至使用。
9. 根据表 10。
重要提示:加热盖必须设置为 85°C 以防止蒸发。表 10. 杂交捕获条件
步 孵化温度 孵化时间 1 95°C 1分钟 2 95°C 5分钟 3 60°C 30 分钟–4 小时* 4 60°C 保持† *少于 30 分钟的杂交时间可能会导致目标区域覆盖率降低。
对热循环仪进行编程,使其在杂交完成后保持在 60°C。不要让杂交反应冷却下来,因为这会对脱靶率产生负面影响。
10. 启动程序。当热循环仪模块达到 95°C 时,暂停步骤 1 中的程序。
11. 涡旋 65°C 快速杂交溶液,并立即将 20 µl 转移到包含步骤 7 中干燥的补充文库池的试管中。
重要提示:快速杂交溶液是粘性的。缓慢移液以确保准确的体积转移。
12. 涡旋管 5 秒,然后在 65°C 下孵育 2 分钟。
13. 再重复步骤 12 2 次,然后脉冲旋转试管。
注意:可能会形成白色沉淀。这不影响杂交过程。
14. 将完整的溶液转移到 0.2 ml PCR 管中并脉冲旋转。确保不存在气泡。
15. 添加 30 µl Vapor-Lock 试剂。脉冲旋转管以确保所有液体都在管的底部。
注意:Vapor-Lock 将在脉冲自旋后在杂交反应的顶部形成一个相。这不影响杂交过程。
16. 将试管转移到热循环仪并在 95°C 下继续程序。
重要提示:确保加热盖设置为 85°C,管密封严密,并且使用了正确的热循环仪盖垫片。
17. 将杂交反应留在热循环仪中,直到准备好将捕获的库片段与链霉亲和素珠结合。在杂交完成前至少 30 分钟继续执行“将杂交目标与链霉亲和素珠结合”的操作步骤。或者,杂交反应可以在热循环仪中保持过夜,以在第二天继续将杂交的靶标与链霉亲和素珠结合。
重要提示:程序完成后,不要让杂交反应冷却至低于 60°C。 -
将杂交靶标与链霉亲和素珠结合
在这一步中,生物素偶联的人类外显子组探针及其杂交靶点将被链霉亲和素珠捕获。在一系列洗涤步骤中,非特异性目标将被洗掉。在杂交完成前至少 30 分钟开始准备 Streptavidin Binding Beads 和洗涤缓冲液。
开始前要做的事情· 将 1.5 ml 试管的加热块预热至 70°C。
· 将 1.5 ml 试管的加热块预热至 48°C。
· 检查捕获后结合缓冲液、洗涤缓冲液 A 和洗涤缓冲液 B 是否有任何可能形成的沉淀物。将缓冲液加热至 48°C 并彻底混合,直至所有沉淀物溶解。
· 将链霉亲和素结合珠平衡至室温至少 30 分钟。
程序
18. 每次反应,分装 450 µl Wash Buffer A 并预热至 70°C。
19. 每次反应,分装 700 µl Wash Buffer B 并预热至 48°C。
注意:将洗涤缓冲液留在加热块中,直到需要为止。
20. 通过涡旋使预平衡的链霉亲和素结合珠均质化。
21. 将 100 µl 均质的链霉亲和素结合珠转移到新的 1.5 ml LoBind 管中。为每个杂交反应准备 1 个试管。
22. 添加 200 µl Post Capture Binding Buffer 并通过移液器混合。
23. 将磁珠在磁力架上孵育 1 分钟或直到溶液澄清,然后取出并丢弃澄清的上清液,不要干扰沉淀。从磁性支架上取下。
24. 重复步骤 22 和 23 2 次以上,总共 3 个洗涤步骤。
25. 从第三次洗涤中去除上清液后,添加最后的 200 µl Post Capture Binding Buffer 并通过涡旋使磁珠均质。将均质的珠浆保持在室温下,直到需要。
26. 杂交反应完成后,打开热循环仪的盖子,打开试管,将包括 Vapor-Lock 在内的全部体积迅速转移到含有步骤 25 中的珠浆的试管中。通过移液器充分混合。
重要提示:在转移到 Streptavidin Binding Beads 之前,不要让杂交反应冷却至低于 60°C。直接从热循环仪快速转移对于最大限度地减少脱靶率至关重要。
27. 在振荡器、旋转器或类似设备上在室温下孵育混合物 30 分钟,以防止珠子沉降。或者,将混合物在室温下孵育 30 分钟,同时每隔 5 分钟搅拌一次,以防止珠子沉降。
注意:不需要剧烈混合。不要涡旋。
28. 脉冲旋转管以确保所有液体都在管的底部。
29. 在磁力架上孵育磁珠 1 分钟或直到溶液澄清,然后取出并丢弃包括 Vapor-Lock 在内的澄清上清液,而不会干扰颗粒。
注意:去除上清液后和每个洗涤步骤中可能会残留少量的 Vapor-Lock。这不会影响最终的捕获产品。
30. 从磁力架上取下试管,加入 200 µl 预热的 70°C 洗涤缓冲液 A。吹打混匀。
31. 在 70°C 下孵育 5 分钟。
32. 将试管放在磁力架上 1 分钟或直到溶液澄清,然后取出并丢弃澄清的上清液,不要干扰颗粒。从磁力架上取下试管,再加入 200 µl 预热的 70°C 洗涤缓冲液 A。吹打混匀。
33. 在 70°C 下孵育 5 分钟。
34. 脉冲旋转试管并将整个体积(包括珠子)转移到新的 1.5 ml LoBind 试管中。
35. 将试管放在磁力架上 1 分钟或直到溶液澄清,然后取出并丢弃澄清的上清液,不要干扰颗粒。
36. 从磁力架上取下试管,加入 200 µl 预热的 48°C 洗涤缓冲液 B。吹打混匀。
37. 脉冲旋转试管,然后在 48°C 下孵育 5 分钟。
38. 将试管放在磁力架上 1 分钟或直到溶液澄清,然后取出并丢弃澄清的上清液,不要干扰颗粒。
39. 重复步骤 36 至 38 2 次以上,总共清洗 3 次。
40. 最后一次洗涤后,使用 10 µl 移液器去除所有残留的上清液。立即进行下一步。
重要提示:不要让珠子变干。
41. 从磁力架上取下试管,加入 45 µl 无 RNase 水。通过移液进行均质化,并将珠浆储存在冰上。
42. 直接进入“捕获后放大”。或者,将珠浆分装成两个 22.5 µl 等分试样,并在 –20°C 下储存以备后用。
重要提示:不要丢弃珠子。捕获产物仍与链霉亲和素珠结合。 -
捕获后放大
与链霉亲和素结合珠结合的捕获目标将使用 QIAGENs Post Hybrid-Capture PCR Mix 进行扩增。扩增的杂交捕获文库将被纯化,并准备好在 Illumina 仪器上进行 QC 和测序。
开始前要做的事情· 准备新鲜的 80% 乙醇。
· 如果储存在 –20°C,将 22.5 µl 的 Streptavidin Binding Bead 浆液在冰上解冻(来自步骤 42)。
· 使用前将 Agencourt AMPure XP 微珠平衡至室温 20-30 分钟。
· 在冰上解冻 Post Hybrid-Capture PCR Mix 和 Primer Mix Illumina Library Amplification。一旦试剂解冻,通过脉冲涡旋混合以避免任何局部浓度。使用前短暂旋转涡旋试剂。
程序
43. 根据表 11。表 11. 捕获后扩增条件
时间 温度 循环次数 2 分钟 98°C 1 20 s 98°C 7 个周期* 30 s 60°C 30 s 72°C 1分钟 72°C 1 ∞ 4°C 抓住 *使用少于 1000 ng 的总捕获池输入时执行 8 个循环。
44. 根据以下方法在冰上制备反应混合物表 12。在 PCR 管或 96 孔 PCR 板中混合成分。表 12. 用于捕获后扩增的反应混合物
零件 体积(微升) 带有捕获目标的珠浆(来自步骤 41) 22.5 杂交捕获后 PCR 混合液 25 Primer Mix Illumina 文库扩增 2.5 总反应体积 50
45. 将 PCR 管或板转移到热循环仪并启动程序。
46. PCR 完成后,将 75 µl 重悬的 Agencourt AMPure XP Beads 添加到每个反应 (50 µl) 中,并上下吸管彻底混合。
47. 在室温下孵育混合物 5 分钟。将磁珠放在磁力架上,小心弃去上清液。
48. 加入 200 µl 80% 乙醇清洗磁珠。将磁珠放在磁力架上,弃去上清液。重复洗涤一次,总共进行 2 次乙醇洗涤。尽可能多地去除多余的乙醇。
49. 在磁力架上孵育 5-10 分钟或直到珠子变干。磁珠过度干燥可能导致 DNA 回收率降低。从磁性支架上取下。
50. 通过重悬于 32 µl 缓冲液 EB、10 mM Tris·Cl、pH 8.0 或无 RNase 的水中进行洗脱。在室温下孵育 2 分钟。
51. 将磁珠放在磁力架上。小心地将 30 µl 透明上清液转移到新试管中。
52. 使用毛细管电泳设备(例如 QIAGEN QIAxcel Advanced 或 Agilent Bioanalyzer 2100)评估文库的质量和大小分布。检查预期的大小分布(参见图 2).为获得最佳测序结果,请使用 qPCR 检测(例如 QIAseq Library Quant Assay Kit 或兼容方法)对捕获的库进行定量。
注意:该库应显示类似于用于相应捕获池的全基因组库的分布。
53. 纯化的文库可以安全地储存在 –20°C 的 DNA LoBind 管中,直到准备好用于测序。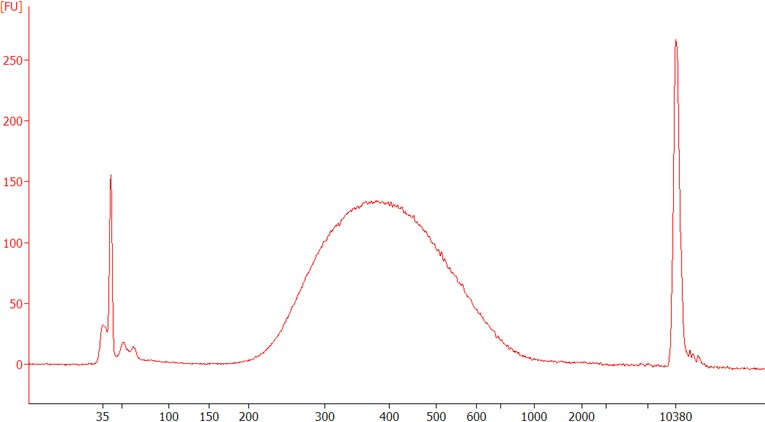
图 2. 最终混合捕获文库库的电泳示踪数据。△点击放大图片
-
测序指南
汇集指南
根据所使用的测序仪器,组合多个杂交捕获池可能对成本有效的测序是可取的。在合并等量的混合捕获池之前,量化每个池并稀释到定义的浓度(例如 2 nM)。要确定单次测序运行中要测序的样本总数,请考虑测序仪器的规格、目标区域大小 (33 Mb) 和每个样本所需的覆盖率。
QIAseq Human Exome Kit 在整个目标区域实现了非常高的覆盖均匀性,典型的 Fold-80 碱基损失在 1.3-1.5 范围内。这意味着平均目标区域覆盖率需要增加大约 1.4,才能将 80% 的非零覆盖目标碱基提高到平均覆盖水平。在规划测序池时,请考虑所需最小覆盖率的大约 1.3 到 1.5 倍,以可靠地获得整个目标区域的覆盖率。
为了计算每次测序运行的整个外显子组库的数量,假设大约整个外显子组覆盖的每 1 倍有 0.11 Gb 的序列输出。
例子:样品: Illumina NextSeq®550,高输出 指定输出: 120 千兆字节 所需的最小覆盖范围: 30x 折叠 80 倍校正覆盖率: 45x [1.5 * 30x] 最大外显子组数: 24 [120 千兆位/(0.11 * 45x)]
测序· 始终确保已使用 QIAseq Library Quant Assay 或兼容的方法对文库进行量化,以在测序池中实现相同的文库表示和准确的池浓度,以实现最佳流动池加载和最佳测序性能。
· 有关如何变性测序文库和设置测序运行的完整说明,请参阅系统特定的 Illumina 文档。
· 根据用于文库制备的适配器类型,请参阅相应的 QIAseq 文库手册,了解有关 QIAseq 组合和独特双标签适配器的详细信息。
· 可编辑且可立即使用的样本表,包括 QIAseq CDI 和 UDI Y-Adapters 的所有样本索引,可从 QIAGEN.com 下载。
· 有关 Illumina 仪器的运行设置以及用于 Illumina Experiment Manager 和 Local Run Manager 的 QIAseq CDI-Y 和 UDI-Y 适配器的定义文件的说明,请访问www.qiagen.com。
· NextSeq、HiSeq X®、HiSeq®3000/4000 系统的测序遵循与其他 Illumina 系统不同的双索引工作流程。如果在这些仪器上手动创建样品表,请输入 i5 索引适配器序列的反向补充。如果使用 Illumina Experiment Manager、BaseSpace®或 Local Run Manager 来计划运行,软件将在必要时自动反转补体索引序列。


