如下操作步骤以产品货号333812的步骤为例,产品列表详见右下方推荐产品
实验步骤
- 1
- 2
- 3
- 4
- 5
- 6
- 7
-
QIAseq 16S/ITS 筛选板
该操作步骤专为放大所有 16S/ITS rRNA 可变区而设计。
开始前的要点
· 使用高质量的 DNA。
· 如果 DNA 浓度 >1 ng/µl,则稀释至 1 ng/µl。如果 DNA 浓度为≤1 ng/µl,然后每次 PCR 反应使用 1 µl 样品。
· 确保反应充分混合,并在推荐的温度下制备和孵育。
· 应使用无核酸酶水新鲜制备 80% 乙醇,并通过涡旋彻底混合。
· QIAseq Beads 需要是同质的。在使用前立即彻底重悬珠子并快速处理珠子。如果操作步骤出现延迟,只需再次涡旋珠子。
程序
1. 在冰上解冻 QIAseq 16S/ITS 筛选板池 1、QIAseq 16S/ITS 筛选板池 2、QIAseq 筛选 16S/ITS 板池 3、UCP 多重预混液、UCP PCR 水和样品。
2. 在冰上,依据表7按照以下步骤为每个 gDNA 样本准备 3 个 PCR 反应。短暂离心,上下吹打混匀 10 次,然后再次短暂离心。表 7. QIAseq 16S/ITS Screening Panel PCR 的制备
零件 面板池 1 面板池 2 面板池 3 微生物 DNA 样本* 1 µl 1 µl 1 µl UCP 多路复用主混音 2. 5 µl 2. 5 µl 2. 5 µl 面板池 1 1 µl – – 面板池 2 – 1 µl – 面板池 3 – – 1 µl UCP PCR 水 5. 5 µl 5. 5 µl 5. 5 µl 总容积 10 µl 10 µl 10 µl *可以添加 QIAseq 16S/ITS Smart Control 代替微生物 DNA 样本。请参阅附录 A:QIAseq 16S/ITS Smart Control 的准备和使用。
3. 如中所述在热循环仪中孵育反应表 8。表 8. QIAseq 16S/ITS PCR 反应的设置
步骤 时间 温度 抓住 2 分钟 95°C 3 步循环 变性 30 s 95°C 退火 30 s 50°C 延伸 2 分钟 72°C 12 个周期* 最终延长(1 个周期) 7 分钟 72°C 抓住 ∞ 4°C *如果样品中细菌/真菌含量低,请使用 20 个循环的 16S PCR。
4. 从热循环仪中取出管/板并短暂离心。
5. 在每个 PCR 反应中加入 20 µl UCP PCR Water。
6. 将来自相同微生物 DNA 样本的 PCR 反应汇集到单个 LoBind 管或 96 孔 PCR 板的孔中。每个微生物 DNA 样本的总体积应为 90 µl。
7. 将 1. 1 倍体积 (99 µl) 的 QIAseq Beads 添加到步骤 5 中的每个组合样品中。通过上下移液 12 次充分混合,然后短暂离心。
8. 在室温下孵育 5 分钟。
9. 将管/板放在磁架上 5 分钟。溶液澄清后,小心取出并丢弃上清液。
10. 短暂离心并小心去除任何残留液体。加入 55 µl 无核酸酶水。通过上下吹打混合 12 次,直到珠子完全重新悬浮。在室温下孵育 2-5 分钟。
注意:此步骤不需要 UCP PCR 水。
11. 将管/板放回磁架直到溶液变清,然后小心地将 50 µl 含有 16S/ITS PCR 产物的上清液转移到新管或板中。
12. 向每个样品中加入 1. 1 倍体积 (55 µl) 的 QIAseq Beads。通过上下吹打 12 次充分混合并短暂离心。
13. 在室温下孵育 5 分钟。
14. 将管/板放在磁架上 5 分钟。溶液澄清后,小心取出并丢弃上清液。
注意:在处理上清液和洗涤步骤时,将磁珠放在磁力架上。
重要提示:不要丢弃磁珠,因为它们含有感兴趣的 DNA。
15. 在磁性架/板上加入 200 μl 80% 乙醇。旋转管子(3 次)或在磁铁的 2 个位置左右移动板以清洗珠子。小心取出并丢弃洗涤液。
16. 重复步骤 15 中的乙醇洗涤。
重要提示:在第二次洗涤后彻底清除所有乙醇洗涤痕迹。为此,请短暂离心并将管或板放回磁力架。首先用 200 µl 移液器除去乙醇,然后使用 10 µl 移液器除去任何残留的乙醇。
17. 管/板仍然在磁铁上,在室温下风干 5 分钟(使用板时最多 15 分钟)。
注意:目视检查颗粒是否完全干燥。当完全干燥时,珠子应该具有“破裂”的外观。
18. 从磁力架上取下试管/板,加入 35 µl UCP PCR 水从磁珠上洗脱 DNA。通过移液充分混合。在室温下孵育 2-5 分钟。
19. 将管/板放回磁架,直到溶液清除。
20. 将 32. 5 µl 上清液转移到干净的试管/板上。
21. 继续“操作步骤:QIAseq 16S/ITS Screening Panel Sample Index PCR Reaction 的制备”,或者,清理后的产品可以储存在 -30 至-15°C 在恒温冰箱中。 -
QIAseq 16S/ITS Screening Panel 样品索引 PCR 反应的制备
开始前的要点
· 在冰上设置 QIAseq 16S/ITS 筛选面板样本索引 PCR 反应。
· 确保反应充分混合,并在推荐的温度下制备和孵育。
· 应使用无核酸酶水新鲜制备 80% 乙醇,并通过涡旋彻底混合。
· QIAseq Beads 需要是同质的。在使用前立即彻底重悬珠子并快速处理珠子。如果操作步骤出现延迟,只需再次涡旋珠子。
程序
1. 在冰上解冻所需的 QIAseq 16S/ITS 指数、UCP Multiplex Master Mix 和 UCP PCR Water。
2. 将含有 16S PCR 产物的管/板放在冰上。
3. 将组件添加到含有 16S PCR 产物的管/板上,根据表 9。如果使用 HT 阵列样品索引格式,请将样品和 UCP Multiplex Master Mix 添加到索引板的相应孔中。短暂离心,上下吹打混合 12 次,然后再次短暂离心。
重要提示:为防止 UCP Multiplex Master Mix 受到污染,将所有样品的足够体积分装到无菌试管中,然后将所需量转移到每个样品中。表 9. QIAseq 16S/ITS 样品索引 PCR 反应的制备
零件 每个样品 HT 阵列(96 或 384 索引套件) 16S PCR 产物 32.5 µl 32.5 µl UCP 多路复用主混音 12.5 µl 12.5 µl p5-RS2-ID# (4 µM)* 2.5 µl - p7-FS2-内径# (4 μM)* 2.5 µl - UCP水 - 5 µl 总容积 50 µl 50 µl *对每个微生物 DNA 样本使用唯一的 p5-RS2-ID# + p7-FS2-ID# 组合。
4. 根据以下步骤在热循环仪中孵育反应表 10。表 10. QIAseq 16S/ITS 样本索引 PCR 反应的设置
步 时间 温度 抓住 2 分钟 95°C 3 步循环 变性 30 s 95°C 退火 30 s 60°C 延伸 2 分钟 72°C 14个周期 最终延长(1 个周期) 7 分钟 72°C 抓住 ∞ 4°C
5. 从热循环仪中取出管/板并短暂离心。
6. 将 0.9X 体积 (45 µl) 的 QIAseq Beads 添加到步骤 5 中的每个样品中。通过上下吹打 12 次充分混合并短暂离心。
7. 在室温下孵育 5 分钟。
8. 将管/板放在磁架上 5 分钟。溶液澄清后,小心取出并丢弃上清液。
注意:在处理上清液和洗涤步骤时,将磁珠放在磁力架上。
重要提示:不要丢弃珠子,因为它们含有感兴趣的 DNA。
9. 在磁性架/板上加入 200 μl 80% 乙醇。旋转管子(3 次)或在磁铁的 2 个位置左右移动板以清洗珠子。小心取出并丢弃洗涤液。
10. 重复乙醇洗涤。
重要提示:在第二次洗涤后彻底清除所有乙醇洗涤痕迹。为此,请短暂离心并将管或板放回磁力架。首先用 200 µl 移液器除去乙醇,然后使用 10 µl 移液器除去任何残留的乙醇。
11. 管/板仍然在磁铁上,在室温下风干 5 分钟(使用板时最多 15 分钟)。
注意:目视检查颗粒是否完全干燥。当完全干燥时,珠子应该具有“破裂”的外观。
12. 从磁力架上取下试管/板,加入 30 µl 无核酸酶水从磁珠中洗脱 DNA。通过移液充分混合。在室温下孵育 2-5 分钟。
注意:此步骤不需要 UCP PCR 水。
13. 将管/板放回磁架,直到溶液清除。
14. 将 25 µl 上清液转移到干净的试管/板上。这是最终的微生物 16S/ITS 筛选板测序库。
15. 继续“操作步骤:文库 QC 和量化”. 或者,完整的 QIAseq 16S/ITS Screening Panel 测序文库可以储存在 -30 至 -15°C 的恒温冰箱中。 -
QIAseq 16S/ITS 区域面板
该操作步骤旨在放大特定的 16S/ITS rRNA 区域。
开始前的要点
· 使用高质量的 DNA。
· 如果 DNA 浓度 >1 ng/µl,则稀释至 1 ng/µl。如果 DNA 浓度为<1 ng/µl,然后每次 PCR 反应使用 1 µl 样品。
· 根据 16S/ITS 区域的数量和数量,16S/ITS 区域 PCR 反应的数量范围为每个样品 1 到 3 个。查看表 11确定每个要设置的样本的 PCR 反应次数。
· 确保反应充分混合,并在推荐的温度下制备和孵育。
· 应使用无核酸酶水新鲜制备 80% 乙醇,并通过涡旋彻底混合。
· QIAseq Beads 需要是同质的。使用前立即彻底重悬珠子,并快速处理珠子。如果操作步骤出现延迟,只需再次涡旋珠子。
程序
1. 如果要对多个 16S 区域进行测序,请使用表 11以确定每个样本的 PCR 反应次数。同时,只有某些区域可以在同一个 PCR 反应中复用在一起。表 11. QIAseq 16S/ITS 区域多路复用指南
V1V2 V2V3 V3V4 V4V5 V5V7 V7V9 它的 V1V2 – X X 是的 是的 是的 是的 V2V3 X – X X 是的 是的 是的 V3V4 X X – X X 是的 是的 V4V5 是的 X X – X X 是的 V5V7 是的 是的 X X – X 是的 V7V9 是的 是的 是的 X X – 是的 它的 是的 是的 是的 是的 是的 是的 – 重要提示:从垂直列中选择一个相关的 16S 区域,然后从水平行中选择一个 16S 区域。如果标记为“是”,则两个区域的引物可以用于同一个 16S PCR 反应(多重)。如果标有“X”,则它们必须在单独的 16S PCR 反应中。计算所需 16S PCR 反应的数量并继续执行步骤 2。
注意:ITS 可以与任何 16S 区域混合使用。
2. 在冰上解冻 QIAseq 16S/ITS Region Panel(s)、UCP Multiplex Master Mix、UCP PCR Water 和样品。
在冰上,按照以下步骤为每个 gDNA 样本准备所需数量的 PCR 反应表 12。短暂离心,上下吹打混合 10 次,然后再次短暂离心。表 12. QIAseq 16S/ITS 区域 PCR 的制备
每个样品 零件 面板池 1 面板池 2(可选) 面板池 3(可选) 微生物 DNA 样本* 1 µl 1 µl 1 µl UCP 主混音 2.5 µl 2.5 µl 2.5 µl 区域面板 1 1 µl 1 µl 1 µl 区域面板 2 可选(1 微升) 可选(1 微升) 可选(1 微升) 区域面板 3 可选(1 微升) 可选(1 微升) 可选(1 微升) UCP PCR 水 变化 变化 变化 总容积 10 µl 10 µl 10 µl *可以添加 QIAseq 16S/ITS Smart Control 代替微生物 DNA 样本。请参阅附录 A:智能控制的准备和使用。
3.如中所述在热循环仪中孵育反应表 13。表 13. QIAseq16S/ITS 区域 PCR 反应的设置
步 时间 温度 抓住 2 分钟 95°C 3 步循环 变性 30 s 95°C 退火 30 s 50°C 延伸 2 分钟 72°C 12 个周期* 最终延长(1 个周期) 7 分钟 72°C 抓住 ∞ 4°C *如果样品中细菌/真菌含量低,请使用 20 个循环的 16S PCR。
4. 从热循环仪中取出管/板并短暂离心。
5. 如果样品需要多次 PCR 反应,则在每个 PCR 反应中加入 20 µl UCP PCR Water,并将来自同一样品的 PCR 反应汇集到单个 LoBind 管或 PCR 板的孔中。如果每个样品只有一个 PCR 反应,添加 40 µl UCP PCR Water。
6. 根据每个样品的 PCR 反应次数,在每个样品中加入 1.1 倍体积的 QIAseq Beads。通过上下吹打 12 次充分混合。
· 每个样品 1 个 PCR 反应 = 每管/孔添加 55 µl QIAseq Beads
· 每个样本 2 个 PCR 反应 = 每管/孔添加 66 µl QIAseq Beads
· 每个样本 3 个 PCR 反应 = 每管/孔添加 99 µl QIAseq Beads
7. 在室温下孵育 5 分钟。
8. 将管/板放在磁架上 5 分钟。溶液澄清后,小心取出并丢弃上清液。
9. 短暂离心并小心去除任何残留液体。
10. 加入 55 µl 无核酸酶水。通过上下吹打混合 12 次,直到珠子完全重新悬浮。在室温下孵育 2-5 分钟。
注意:此步骤不需要 UCP PCR 水。
11. 将管/板放回磁架直到溶液变清,然后小心地将 50 µl 含有 16S/ITS PCR 产物的上清液转移到新管或板中。
12. 向每个样品中加入 1.1 倍体积 (55 µl) 的 QIAseq Beads。通过上下吹打 12 次充分混合。
13. 在室温下孵育 5 分钟。
14. 将管/板放在磁架上 5 分钟。溶液澄清后,小心取出并丢弃上清液。
注意:在处理上清液和洗涤步骤时,将磁珠放在磁力架上。
重要提示:不要丢弃磁珠,因为它们含有感兴趣的 DNA。
15. 在磁性架/板上加入 200 μl 80% 乙醇。旋转管子(3 次)或在磁铁的 2 个位置左右移动板以清洗珠子。小心取出并丢弃洗涤液。
16. 重复乙醇洗涤。
重要提示:在第二次洗涤后彻底清除所有乙醇洗涤痕迹。为此,请短暂离心,然后将管或板放回磁力架。首先用 200 µl 移液器除去乙醇,然后使用 10 µl 移液器除去任何残留的乙醇。
17. 管/板仍然在磁铁上,在室温下风干 5 分钟(使用板时最多 15 分钟)。
注意:目视检查颗粒是否完全干燥。当完全干燥时,珠子应该具有“破裂”的外观。
18. 从磁力架上取下试管/板,加入 35 µl UCP PCR 水从磁珠上洗脱 DNA。通过移液充分混合。在室温下孵育 2-5 分钟。
19. 将管/板放回磁架,直到溶液清除。
20. 将 32.5 µl 上清液转移到干净的试管/板上。
21. 继续“操作步骤:QIAseq 16S/ITS Region Panel Sample Index PCR Reaction 的制备”,或者,净化后的 PCR 产物可以储存在 -30 至-15°C 在恒温冰箱中。 -
QIAseq 16S/ITS Region Panel Sample Index PCR 反应的制备
开始前的要点
· 在冰上设置 QIAseq 16S/ITS 区域面板样本索引 PCR 反应。
· 确保反应充分混合,并在推荐的温度下制备和孵育。
· 应使用无核酸酶水新鲜制备 80% 乙醇。
· QIAseq Beads 需要是同质的。使用前立即彻底重悬珠子,并快速处理珠子。如果操作步骤出现延迟,只需再次涡旋珠子。
程序
1. 解冻所需的 QIAseq 16S/ITS 指数、UCP Multiplex Master Mix 和 UCP PCRWater。
2. 将含有 QIAseq 16S/ITS Region Panel PCR 产物的管/板置于冰上。
3. 将组件添加到含有 16S PCR 产物的管/板上,根据表 14。如果使用 HT 阵列样品索引格式,请将样品和 UCP Multiplex Master Mix 添加到索引板的相应孔中。短暂离心,上下吹打混合 12 次,然后再次短暂离心。
重要提示:为防止低生物负载预混液受到污染,将足够体积的所有样品分装到无菌试管中,然后将所需量转移到每个样品中。表 14. QIAseq 16S/ITS Region Panel Sample Index PCR 反应的制备
步 每个样品 HT 阵列(96 或 384 索引套件) 16S/ITS 区域面板 PCR 产物 32.5 µl 32.5 µl UCP 主混音 12.5 µl 12.5 µl p5-RS2-ID# (4 µM)* 2.5 µl – p7-FS2-内径# (4 μM)* 2.5 µl – UCP水 – 5 µl 总容积 50 µl 50 µl *对每个微生物 DNA 样本使用唯一的 p5-RS2-ID# + p7-FS2-ID# 组合。
4. 根据以下步骤在热循环仪中孵育反应表 15。表 15. QIAseq 16S/ITS Region Panel Sample Index PCR 反应的制备
步 时间 温度 抓住 2 分钟 95°C 3 步循环 变性 30 s 95°C 退火 30 s 60°C 延伸 2 分钟 72°C 19 个周期 最终延长(1 个周期) 7 分钟 72°C 抓住 ∞ 4°C
5. 从热循环仪中取出管/板并短暂离心。
6. 将 0.9 倍体积 (45 µl) 的 QIAseq Beads 添加到步骤 5 中的每个样品中。通过上下吹打 12 次充分混合。
7. 在室温下孵育 5 分钟。
8. 将管/板放在磁架上 5 分钟。溶液澄清后,小心取出并丢弃上清液。
注意:在处理上清液和洗涤步骤时,将磁珠放在磁力架上。
重要提示:不要丢弃磁珠,因为它们含有感兴趣的 DNA。
9. 在磁性架/板上加入 200 μl 80% 乙醇。旋转管子(3 次)或在磁铁的 2 个位置左右移动板以清洗珠子。小心取出并丢弃洗涤液。
10. 重复乙醇洗涤。
重要提示:在第二次洗涤后彻底清除所有乙醇洗涤痕迹。为此,请短暂离心并将管或板放回磁力架。首先用 200 µl 移液器除去乙醇,然后用 10 µl 移液器除去任何残留的乙醇。
11. 管/板仍然在磁铁上,在室温下风干 5 分钟(使用板时最多 15 分钟)。
注意:目视检查颗粒是否完全干燥。当完全干燥时,珠子应该具有“破裂”的外观。
12. 从磁力架上取下试管/板,加入 30 µl 无核酸酶水从磁珠中洗脱 DNA。通过移液充分混合。在室温下孵育 2-5 分钟。
注意:此步骤不需要 UCP PCR 水。
13. 将管/板放回磁架,直到溶液清除。
14. 将 25 µl 上清液转移到干净的试管/板上。这是最终的 16S/ITS 区域面板测序库。
15. 继续“操作步骤:图书馆 QC 和量化”,或者,完整的 QIAseq 16S/ITS 区域面板测序库可以存储在 -30 至-15°C 在恒温冰箱中。 -
文库 QC 和量化
开始前的要点
· QIAseq 16S/ITS Screening Panel 或 QIAseq 16S/ITS Region Panel 的一部分,总体积为 25 µl 的测序文库,是文库 QC 和量化的起始材料。不使用时,QIAseq 16S/ITS Screening 或 Region Panel 测序文库应存放在冰上。
· 文库 QC 涉及使用 Agilent 2100 生物分析仪或 TapeStation。
· 文库量化涉及使用 QIAGEN 的 QIAseq Library Quant System:QIAseq Library Quant Array Kit(货号 333304)或 QIAseq Library Quant Assay Kit(货号 333314)。
文库 QC(Agilent 2100 生物分析仪或 TapeStation)
1. 根据制造商的说明,使用高灵敏度 DNA 芯片在安捷伦生物分析仪或 TapeStation 上分析 1 µl QIAseq 16S/ITS Screening Panel 或 QIAseq 16S/ITS Region Panel 测序文库。典型的 QIAseq 16S/ITS Screening Panel 或 QIAseq 16S/ITS Region Panel 测序文库显示在图 2。图 3显示了一个由 QIAseq 16S/ITS Smart Control 构建的文库。这些文库有一个额外的约 440 个碱基对峰,因为这代表 ITS 扩增子,其丰度与其他 16S 扩增子相同。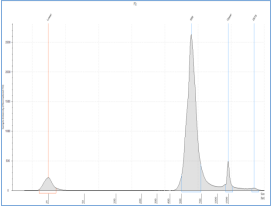
图 2. 使用 QIAseq 16S/ITS Screening Panel 或 QIAseq 16S/ITS Region Panel 制备的文库的生物分析仪轨迹。△点击放大图片

图 3. 使用 QIAseq 16S/ITS Smart Control(作为 DNA 样本)和 QIAseq 16S/ITS Screening Panel 制备的文库的生物分析仪轨迹。△点击放大图片
2. 进入下一页的“文库量化”。
文库量化
3. Bioanalyzer 或 TapeStation 系统的文库产量测量使用嵌入 DNA 或 RNA 的荧光染料,无法区分带有或不带有接头序列的 DNA。基于实时荧光定量 PCR 的方法可准确定量完整的 QIAseq 16S/ITS Screening Panel 或具有完整接头序列的 QIAseq 16S/ITS Region Panel 测序文库。因此,强烈建议使用 QIAGEN 的 QIAseq Library Quant Array Kit 或 Assay Kit,其中包含实验室验证的正向和反向引物以及 DNA 标准品,以准确定量制备的文库。
2 nM 的 QIAseq 16S/ITS Screening Panel 或 QIAseq 16S/ITS Region Panel 文库应用作加载 MiSeq 测序仪器的变性程序的输入。
4. 继续“操作步骤:Illumina MiSeq 上的测序设置”, -
Illumina MiSeq 上的测序设置
开始前的要点
· 文库稀释浓度和文库加载浓度的建议基于 QIAGEN 的 QIAseq Library Quant System(参见“操作步骤:文库 QC 和量化”).详情可在QIAGEN官网下载货号为:333812的完整说明书进行了解。
· 重要提示:在 Illumina 平台上进行测序时,必须使用 QIAseq Read1 Primer (Custom Read1 Sequencing Primer) 和 QIAseq 16S/ITS Read 2 Primer (Custom Read2 Sequencing Primer)。
· 定制读取引物进入以下特定的 MiSeq 试剂盒位置:
· QIAseq Read1 Primer: MiSeq Position #18
· QIAseq 16S/ITS 读取 2 引物:MiSeq 位置 #20
· Illumina 平台上的 QIAseq 16S/ITS Screening Panel 或 QIAseq 16S/ITS Region Panel 必须使用双末端测序。
· 有关如何使测序文库变性、准备自定义索引引物和设置测序运行的完整说明,请参阅系统特定的 Illumina 文档。
MiSeq 的测序准备工作
1. 样品表设置:
使用 Illumina Experiment Manager v1.2 设置带有 Custom Sequencing Read 1 Primer 和 Custom Sequencing Read 2 Primer 的样品表。
注意:如果使用 Illumina Experiment Manager v1.4 或更高版本,请参阅附录 B:使用 Illumina Experiment Manager 的更新版本进行设置。
QIAseq 16S/ITS Screening Panel 或 QIAseq 16S/ITS Region Panel 样本索引与 Illumina 的 TruSeq HT 适配器样本索引系统兼容。
选择并检查参数如下(图 4a 和 4b):
· 类别:其他
· 选择应用程序:仅限 FASTQ
· 文库制备试剂盒:TruSeq HT(384-index 试剂盒使用 Nextera XT v2)
· 索引读取:2
· 读取类型:双端
· 读取 1 的循环数:276(如果使用 MiSeq v2 500 循环试剂盒,则为 251)
· 读取 2 的循环数:276(如果使用 MiSeq v2 500 循环试剂盒,则为 251)
· 重要提示:选中“读取 1 的自定义引物”
· 重要提示:选中“读取 2 的自定义引物”
· 重要提示:选中“使用适配器微调”
· 重要提示:选中“使用适配器微调读取 2”
图 4a.使用 Illumina 实验管理器的样品表向导。 MiSeq 应用程序选择。△点击放大图片

图 4b.使用 Illumina 实验管理器的样品表向导。工作流参数。△点击放大图片
2. 样品稀释和汇集:
将 MiSeq 的最终库稀释至 2 nM。
然后,如果每个库需要相似的测序深度,则以等摩尔量将具有不同样本索引的库组合起来。
注意:文库稀释浓度建议基于 QIAseq Library Quant System。
3. 文库准备和加载:
根据 MiSeq 系统变性和稀释文库指南 (sapac.support.illumina.com/downloads/prepare_libraries_for_sequencing_miseq_15039740.html) 在 MiSeq 上准备和加载文库。
在 MiSeq(v3 试剂盒)上,最终变性文库浓度为 10 pM 或 6 pM(v2 试剂盒)。
注意:关于文库加载的建议基于 QIAseq Library Quant System。
4. 用于读取 1 和读取 2 准备和加载的定制测序引物:
使用 597 µl HT1(杂交缓冲液)稀释 3 µl QIAseq Read1 Primer 以获得 0.5 µM 的最终浓度。
使用 597 µl HT1 稀释 3 µl QIAseq Read2 Primer 以获得最终浓度0.5 µM.
将 600 µl 稀释的 QIAseq Read1 Primer 加载到位置 #18,并将 600 µl 稀释的 QIAseq Read2 Primer 加载到 MiSeq 试剂盒的 #20 位置。
有关更多详细信息,请参阅 Illumina 的 MiSeq 系统:自定义引物指南 (sapac.support.illumina.com/downloads/miseq-system-custom-primers-guide-15041638.html)。有关将引物加载到 MiSeq 试剂盒上的位置的指导,请参阅图 5。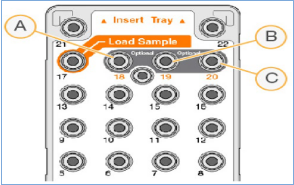
图 5. 将引物加载到 MiSeq 试剂盒上的位置。 (A) Read1 自定义底漆的第 18 位。
(B) 未使用。 (C) Read2 自定义底漆的位置 20。△点击放大图片
5. 完成测序运行后,继续“操作步骤:使用 CLC 微生物基因组学模块进行数据分析”, -
使用 CLC 微生物基因组学模块进行数据分析
开始前的要点
· 如果使用 CLC 微生物基因组学模块,请参阅 CLC 微生物基因组学模块用户手册 (resources.qiagenbioinformatics.com/manuals/clcmgm/current/User_Manual.pdf)。
细菌和真菌分类
1. 导入 FASTQ 文件。
导入 → Illumina→ 检查配对读取 → 选择 FASTQ 文件
2. 解复用扩增子(用于筛选面板或多个区域面板)。
Toolbox → QIAGEN 16S Panel Analysis → Demultiplex Reads by Barcode → 如果解复用多个样本,检查 Batch → Select FASTQ files → 在元数据表中,选择条形码所有文件;在条形码中,选择条形码 → 选中保存
3. 细菌分类
工具箱 → 微生物基因组学模块 → 宏基因组学 → 基于扩增子的 OTU 聚类 → 工作流程 → 数据 QC 和 OTU 聚类 → 如果使用筛选面板或多个区域面板,请为所有样本选择相同的区域(即样本 1-24:V1V2)→ 质量限制= 0.05(默认)→ OTU 拣货 = 基于参考的 OTU → 选择 OTU 数据库 → 检查保存
如果适用,请为每个区域重复。
在 OTU 聚类分析之后,如果需要,可以将 OTU 表合并为一张表。
工具箱®微生物基因组学模块®宏基因组学®丰度分析®合并丰度表
4. 真菌分类
工具箱 → 微生物基因组学模块 → 宏基因组学 → 基于扩增子的 OTU 聚类 → 工作流程 → 数据 QC 和 OTU 聚类 → 选择多路分解的 FASTQ 文件。为每个样品选择 ITS 区域(即样品 1-24,ITS 区域)→ 质量限制 =
0.05(默认)→ OTU 拾取 = 基于参考的 OTU → 选择 UNITE → 检查保存
5. 比较 16S 可变区——比较不同 16S 可变区在多样性方面的表现。
要确定哪个区域包含最多的 OTU 序列,因此可能具有最高的多样性,请检查每个区域分析的 OTU 报告中的“总预测 OTU”。
或者,使用步骤 3 中的合并丰度表(在表视图中),选择“聚合样本”下的区域。然后选择包含在目标分类级别(即科、属或种)上大于 5-10 个读数的特征数量最多的区域。此外,可以测量 alpha 多样性以确定哪个区域表现出最高的多样性。
6. 如需更深入的分析,请参阅 CLC 微生物基因组学模块用户手册。


