设计引物也有多个软件可以使用,这里介绍的是常用的Primer 5软件。
设计引物的基本原则是:
1)引物长度一般为15-30bp,常用的为18-27bp,但不能大于38bp;
2)引物GC含量一般为40%-60%,以45-55%为宜,上下游引物GC含量和Tm值要保持接近;
3)引物所对应的模板序列的Tm值最好在72℃左右;
4)3'端最好不要是连续碱基,GGG或CCC会导致错误的引发,同时3'端最后一个碱基最好不要是A或T,否则容易导致错配;
5)以公式Tm=4*(G+C)+2*(A+T)-5计算Tm值,也就是退火温度。选择较低Tm值的引物的退火温度为反应的退火温度,最好保证每个引物的Tm值相匹配,且在70-75℃范围内;
6)在DNA测序和PCR中最好用5'末端稳定(GC含量多),而3'端不稳定(AT含量多)的引物,这种引物的结构可以有效地消除假引发反应;
7)引物和产物之间的Tm值相差别太大,20摄氏度范围内最好。
此外,在引物的两端需要加上酶切位点和保护碱基。
如果载体上没有标签序列,还需要在引物设计时加上标签序列。
以下对步骤进行梳理,详细操作请查看视频。
实验步骤
- 1
- 2
- 3
- 4
-
输入目标序列
打开Prime 5软件,点击File—New—DNA Sequence
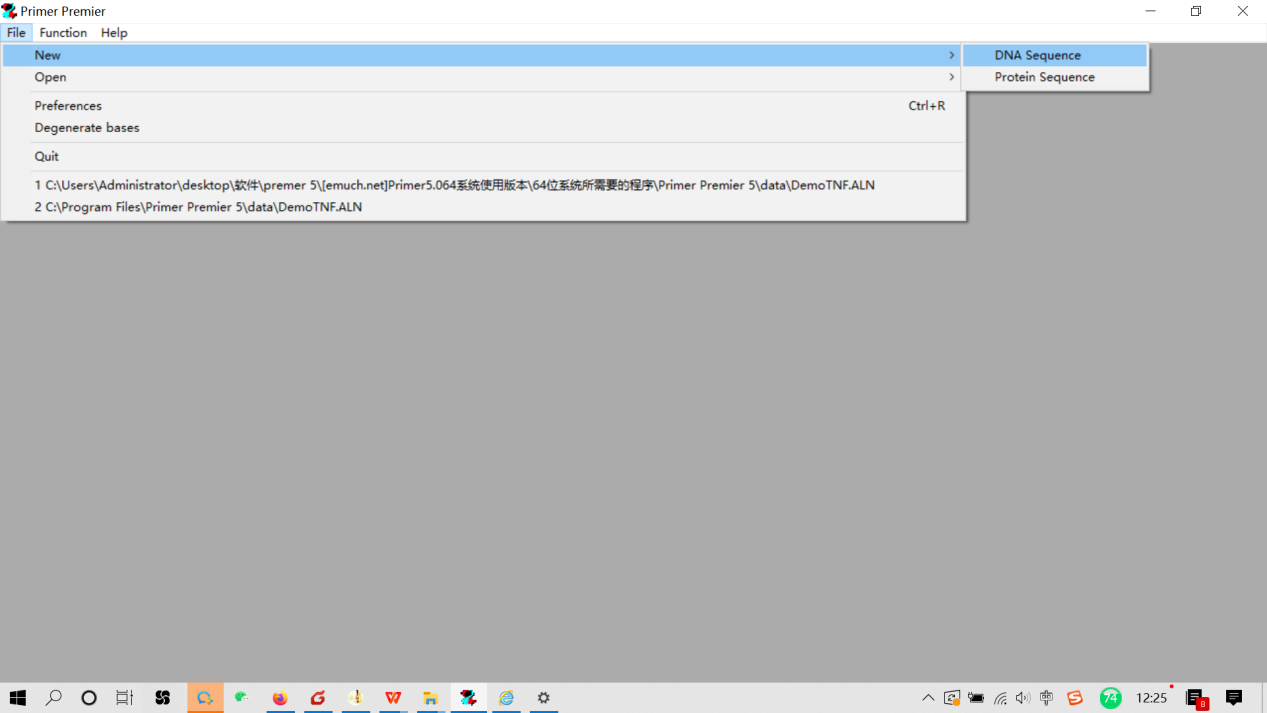
△点击放大图片
点击空白处粘贴我们之前保存的目的基因的CDS序列,选择As is。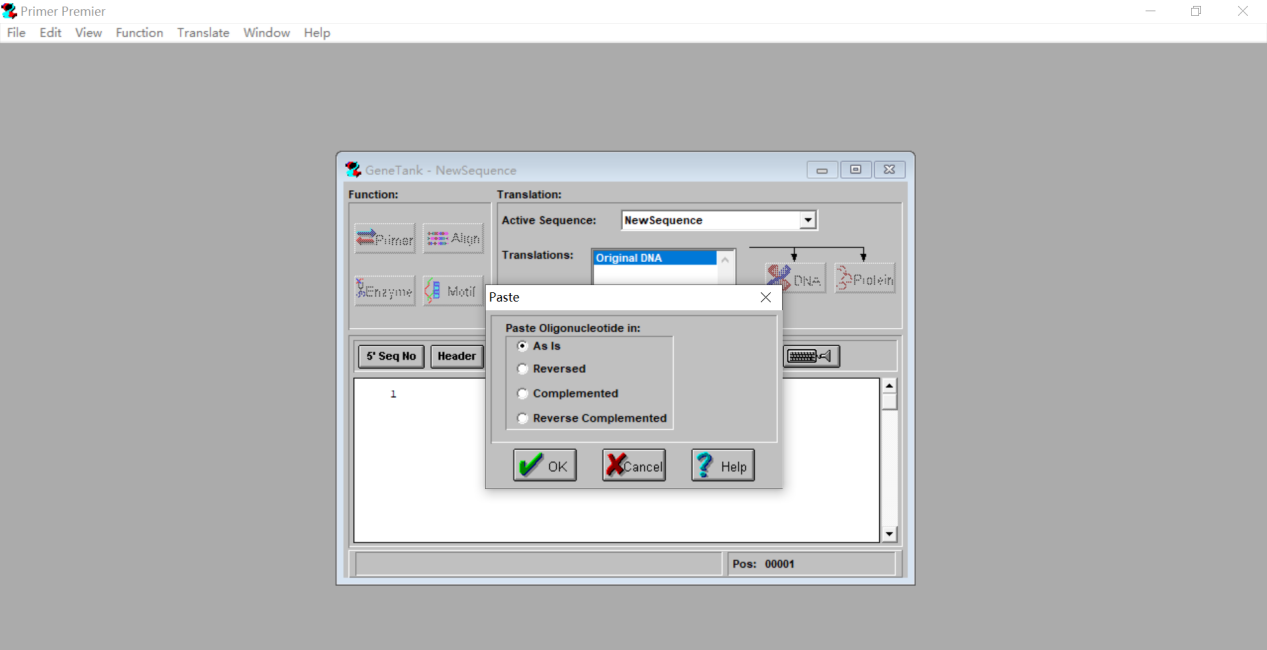
△点击放大图片

△点击放大图片
-
设计引物
点击左上角的Primer,S为正向引物,A为反向引物

△点击放大图片

△点击放大图片
-
对引物进行编辑和调整
点击Edit Primers编辑序列,可调整引物长短,添加酶切位点序列以及保护碱基。如果显示Hairpin,Dimer,False Priming,Cross Dimer或者Tm值太高或太低,则需要调整。

△点击放大图片
-
检测引物特异性
NCBI主页最下端找到Primer-BLAST,打开;

△点击放大图片
输入上下游引物序列,点击最下方
△点击放大图片
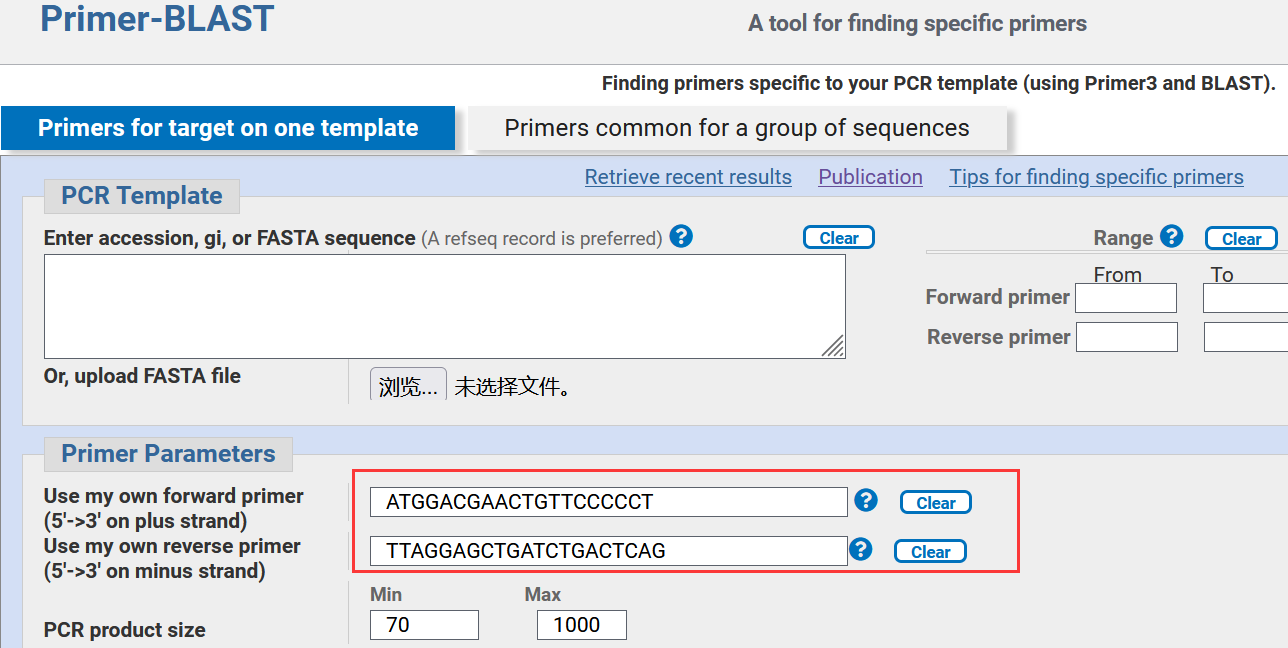
△点击放大图片
如果只能检测到你的目的蛋白基因,则表明特异性较好。


